Clusteranalyse
Skript mit Erklärung der Verfahren und Vorgehensweisen anhand von Beispielen.
Hierarchische und partitionierende Clusteranalyse
Distanzmatrix
Die Distanzmatrix ist ein wichtiger Ausgangspunkt für die Clusteranalyse. Ihre Berechnung hängt vom Skalenniveau der berücksichtigten Variablen ab.
Mit der Funktion daisy() des Pakets “cluster” kann eine Distanzmatrix von Variablen mit unterschiedlichen Skalenniveaus berechnet werden. Wenn in einer Zeile nicht alle Messwerte fehlen, müssen Sie kein listwise deletion durchführen, da bei der Distanzberechnung das Gewicht eines Vergleichs 0 gesetzt wird, sobald Missings vorhanden sind, was zu weniger Datenverlust führt. Deshalb verwenden wir für die Distanzberechnung mit daisy() den vollständigen Datensatz (data2).
Wenn alle Variablen intervallskaliert sind, können Sie die Distanzmatrix wie folgt berechnen: d <- daisy(data2, metric=“euclidean”, stand=TRUE). Mit “stand=TRUE” verlangen Sie, dass die Variablen zuerst standardisiert werden: (Messwert - Mittelwert)/(Mittelwert der absoluten Abweichungen).
Wenn alle Variablen dichotom sind, können Sie mit der Funktion dist.binary() des Pakets “ade4” zwischen 10 verschiedenen Ähnlichkeitsmassen s wählen. Beachten Sie dass s mit der Transformation sqrt(1-s) in Unähnlichkeiten umgerechnet wird. Ein Nachteil dieser Variante ist, dass für alle Variablen dasselbe Ähnlichkeitsmass berechnet wird. Diesen Nachteil können Sie beheben, wenn Sie die Distanzmatrix mit daisy() berechnen. Allerdings können Sie nur zwischen zwei Varianten wählen, vgl. nächster Abschnitt. Wenn Sie mit dist.binary() arbeiten, müssen die Missings zuerst mit listwise deletion entfernt werden (data3).
Wenn die Variablen unterschiedliche Skalenniveaus aufweisen, muss man die Funktion daisy() des Pakets “cluster” verwenden. Intervallskalierte Variablen müssen in daisy() nicht unter type definiert werden. Sie werden wie folgt auf das Intervall [0,1] normiert: (Messwert - Minimum)/Range. Beachten Sie, dass der Range stark von Ausreissern abhängen kann. Wenn Sie in der Funktion daisy() eine Vatiable als type “ordratio” definieren, wird sie intern in eine Variable der Klasse “ordered factor” umgewandelt. Faktoren müssen im Datensatz als Variablen der Klasse “factor” enthalten sein und müssen in daisy() nicht unter type spezifiziert werden. Dichotome Variablen sollten als Faktoren mit den Stufen “1” für Merkmal vorhanden und “0” für Mekrmal nicht vorhanden kodiert werden. Ein Vorteil von daisy() ist, dass Sie im selben Datenfile dichotome Variablen mit unterschiedlicher Behandlung der Übereinstimmung 0-0 haben können (vgl. Beispiel). Definiert man in daisy() eine dichotome Variable als type “asymm”, wird im Fall, dass in zwei Zeilen bei dieser Variable den Wert 0 steht, das Gewicht für diesen Vergleich auf 0 gesetzt. Dies führt bei der Berechnung der Distanz zu einem höheren Wert, weil dadurch die Summe der Gewichte kleiner wird (vgl. Formel zur Berechnung der Distanzen). Definiert man in daisy() eine dichotome Variable als type “symm”, erhält auch eine 0-0-Differenz das Gewicht 1, was im Vergleich zu type “asymm” eine kleinere Distanz ergibt. Faustregel: Wenn “0” eine eindeutige inhaltliche Bedeutung hat, spricht dies für type “symm” (z.B. “1”=Symptom vorhanden vs. “0”=Symptom nicht vorhanden). Wenn “0” keine eindeutige inhaltliche Bedeutung hat, spricht dies für type “asymm” (z.B. “1”=Inländer vs. “0”=Ausländer).
In unseren Beispielen werden die Clusteranalysen anhand der Distanzmatrix gerechnet.
Erforderliche Pakete laden
library(ade4) # Distanzen, binäre Variablen (dist.binary)
library(cluster) # Distanzen, unterschiedliche Skalenniveaus (daisy), Partitioning Around Medoids (pam)
library(fpc) # Vergleich zweier Clusterlösungen (cluster.stats), Stabilität (clusterboot)Hierarchisch agglomerative Clusteranalyse
Datensatz einlesen, Variablen und Aglomerationsmethode spezifizieren, Distanzmatrix berechnen
# Dateneingabe
data(plantTraits) # Mit ?plantTraits erhalten Sie die Variablenbeschreibungen
data <- plantTraits # Lassen Sie sich mit str(data) die Variablenklassen anzeigen.
# Zu verwendende Variablen
Variablen <- 1:31 # Spaltennummern der zu berücksichtigenden Variablen
Namen <- NULL # Spaltennummer mit den Zeilennamen. Verwenden der vorhandenen Zeilennamen: Namen <- NULL
# Aglomerationsmethode wählen, vgl. ?agnes
Methode <- "average" # "average", "single", "complete", "ward", "weighted", "gaverage"
# Distanzmatrix berechnen
# Wenn alle Variablen dasselbe Skalenniveau haben, kann unter "metric" zwischen "euclidean", manhattan"
# oder "gover" gewählt werden.
# Wenn die Variablen unterschiedliche Skalenniveaus aufweisen, kann man diese unter "type" spezifizieren
# ("ordratio"=ordinal, "asymm"=asymmetrisch binär und "symm" symmetrisch binär). Intervallskalierte und
# nominale Variablen werden automatisch erkannt und müssen nicht deklariert werden.
# Beispiel: d <- daisy(data2, type=list(ordratio = 3:5, symm = 9:10, asymm = 11:12))
# Diese Spaltennummern beziehen sich auf data2. Die Spalten 6:7 könnten intervallskalierte Variablen und
# die Spalte 8 eine nominale Variable mit 4 Stufen enthalten.
library(cluster); library(ade4)
if (!is.null(Namen)) rownames(data) <- data[,Namen] # Fallnamen als Zeilennamen definieren
data2 <- data[,Variablen, drop=FALSE]
data2 <- data2[rowSums(is.na(data2))!=ncol(data2), ] # Fälle ausschliessen, bei denen alle Werte fehlen
d <- daisy(data2, type=list(ordratio=4:11, symm=12:13, asymm=14:31)) # Spaltennummern von Data2!Clusteranalyse
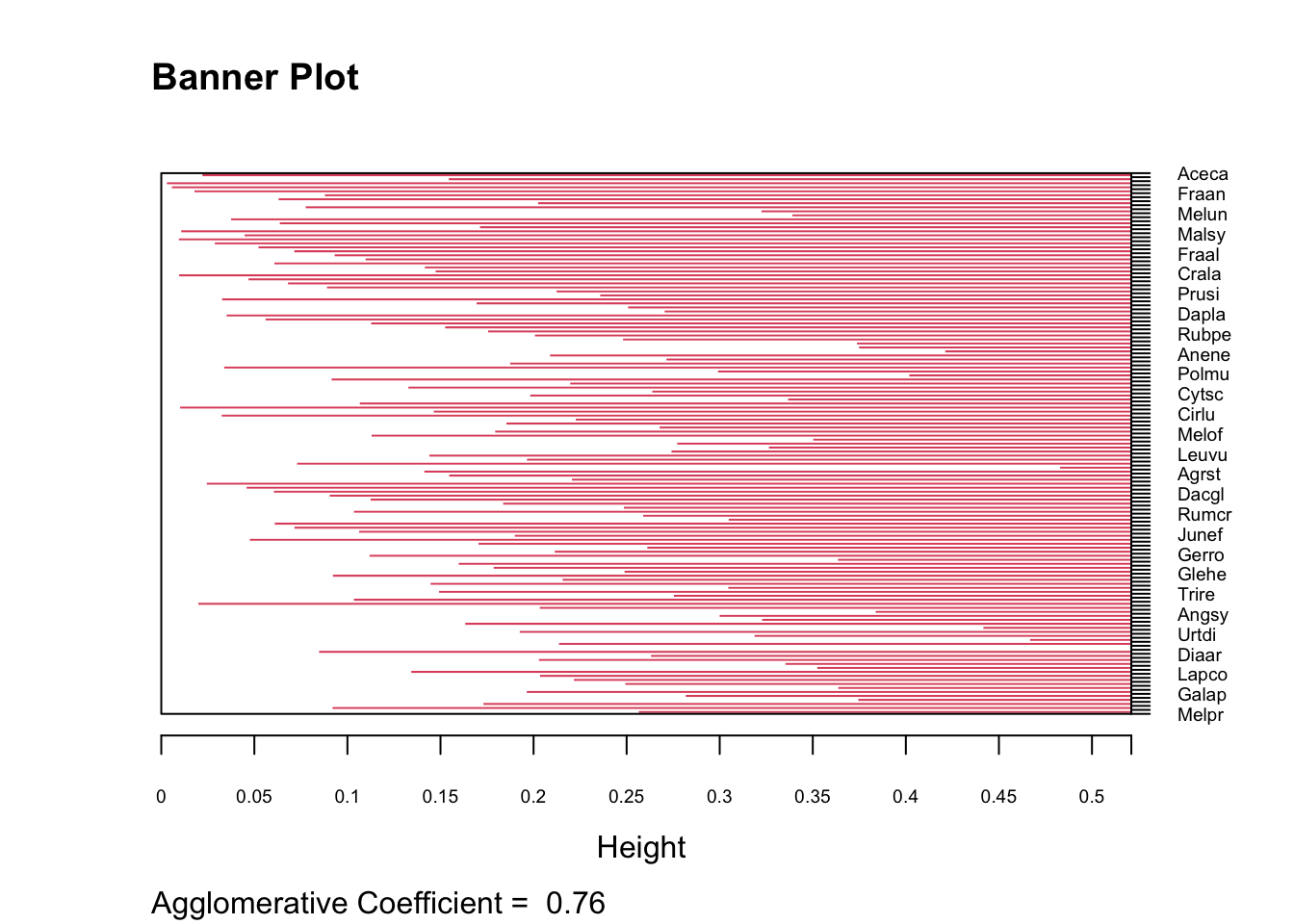
res <- agnes(d, method=Methode)
res## Call: agnes(x = d, method = Methode)
## Agglomerative coefficient: 0.756042
## Order of objects:
## [1] Aceca Aceps Betsp Carbe Frasp Fraan Poptr Ulmsp Corav Fagsy Melun Brydi
## [13] Cucba Tamco Corsa Malsy Pruav Sordo Sorto Pruce Fraal Evoeu Soldu Symsp
## [25] Samni Crala Cramo Rosar Rosca Pyrsp Prusi Ligvu Lonpe Rubfr Rubca Dapla
## [37] Launo Prula Ileaq Rusac Rubpe Hedhe Taxba Queil Robps Anene Conmj Aruma
## [49] Hyahi Hyano Polmu Artvu Holla Potre Teusc Cytsc Eupam Calse Linre Scrno
## [61] Cirlu Stasy Cirar Hyppe Galmo Melof Vicse Calvu Ulemi Epipa Leuvu Verch
## [73] Steho Vinmi Agrca Agrst Milef Cardi Poane Rumsa Dacgl Poatr Poapr Molca
## [85] Meran Rumcr Rumac Antod Junco Elyca Junef Brasy Desfl Holmo Dauca Gerro
## [97] Geuur Ajure Chema Luzmu Glehe Potst Ranac Ranre Frave Trire Privu Viohi
## [109] Viore Tarsp Angsy Rumco Cirvu Digpu Arrel Urtdi Plama Polin Pteaq Allpe
## [121] Diaar Lacse Sonol Paprh Anaar Lapco Poaan Steme Medlu Brost Galap Vicsa
## [133] Cheal Cordi Polav Melpr
## Height (summary):
## Min. 1st Qu. Median Mean 3rd Qu. Max.
## 0.002521 0.087939 0.175070 0.182131 0.261597 0.521138
##
## Available components:
## [1] "order" "height" "ac" "merge" "diss" "call"
## [7] "method" "order.lab"Baumdiagramm für eher kleine Fallzahlen
plot(res, which.plots=2, main="Baumdiagramm", xlab="", sub="", cex=0.6)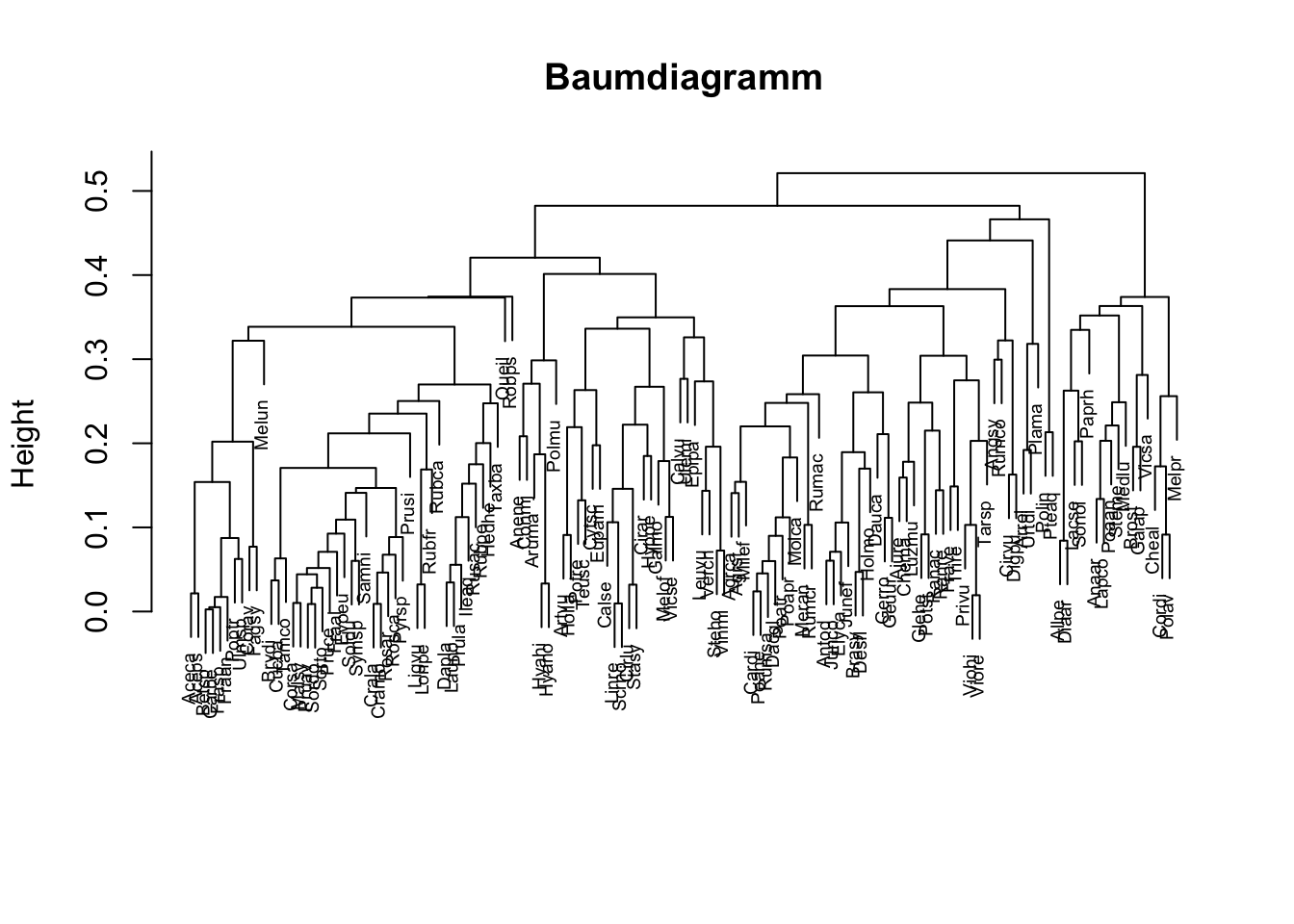
Scree Plot für hohe Fallzahlen
# Es wird das Kriterium "height" in Abhängigkeit der letzten 10 Fusionierungsschritte dargestellt.
height <- sort(res$height)
Schritt <- (length(height)-9):length(height)
height <- height[(length(height)-9):length(height)]
data4 <- data.frame(Schritt, height)
library(ggplot2)
ggplot(data4, aes(x=Schritt, y=height)) + geom_line() +
scale_x_continuous(breaks=Schritt)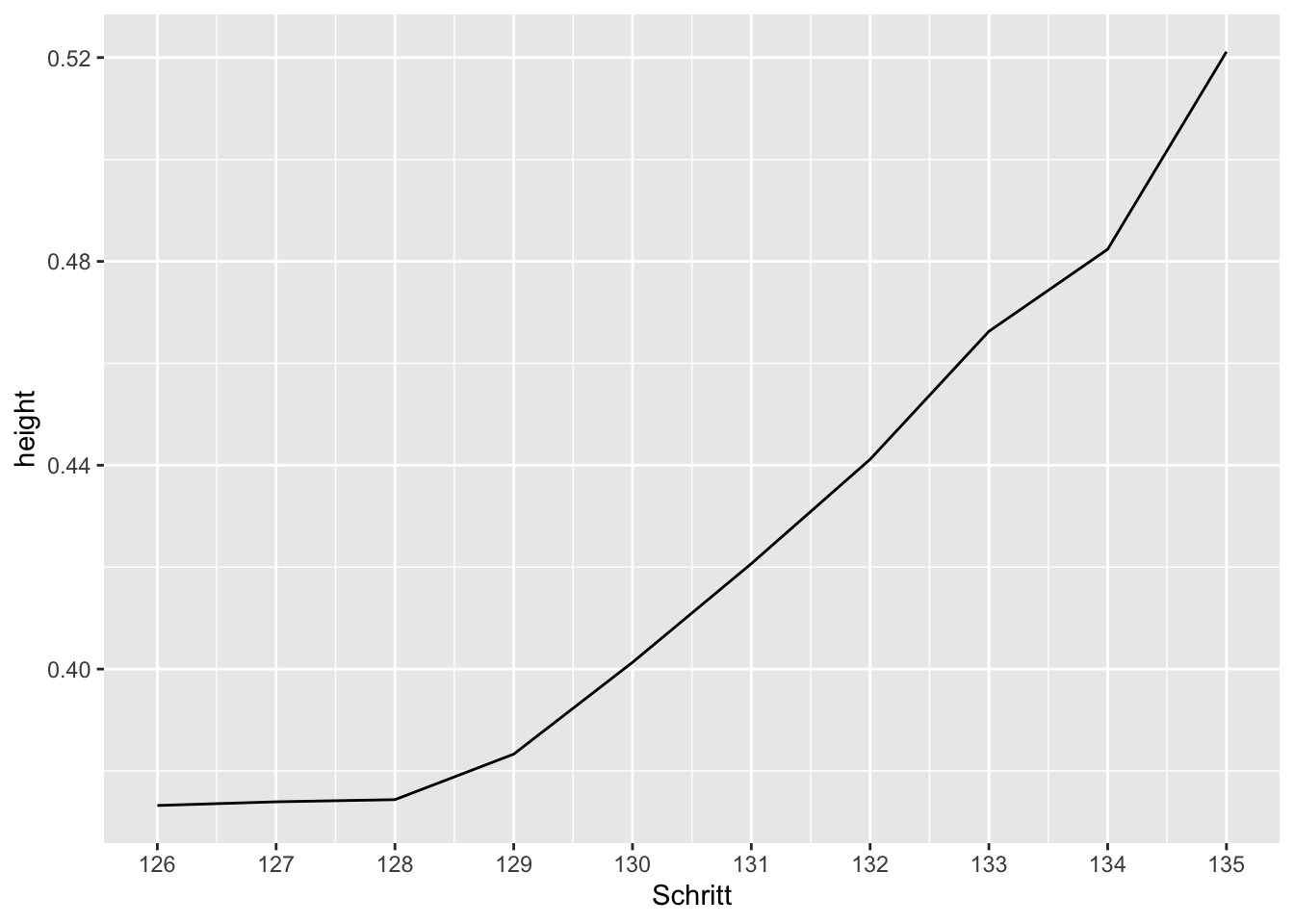
Überprüfung der Stabilität mit Bootstrap
k = 6 # Anzahl Cluster eingeben
library(fpc)
stab <- clusterboot(d, B=100, distances=TRUE, bootmethod="boot", clustermethod=hclustCBI, k=k, cut="number", method=Methode, showplots=FALSE)
stab # Interpretation vgl. ?clusterbootstab## * Cluster stability assessment *
## Cluster method: hclust/cutree
## Full clustering results are given as parameter result
## of the clusterboot object, which also provides further statistics
## of the resampling results.
## Number of resampling runs: 100
##
## Number of clusters found in data: 6
##
## Clusterwise Jaccard bootstrap (omitting multiple points) mean:
## [1] 0.9023781 0.8191123 0.8363167 0.6579700 0.5724174 0.6250000
## dissolved:
## [1] 0 3 6 18 51 39
## recovered:
## [1] 92 72 75 37 24 61
Hinweis 1: Variable mit der Clusterzugehörigkeit ins Datenfile einfügen
# Vektor "Cluster" mit Clusterzugehörigkeit
# Cluster <- cutree(as.hclust(res), k = 6) # Clusteranzahl k anpassen
# Vektor "Cluster" ins Datenfile "data" einfügen
# dat <- numeric(length=nrow(data)); dat[dat==0] <- NA
# dat[rownames(data) %in% names(Cluster)] <- Cluster
# data$Cluster <- dat
Hinweis 2: Baumdiagramm mit markierten Clustern für eine vorgegebene Clusteranzahl k
plot(res, which.plots=2, main="Baumdiagramm", xlab="", sub="", cex=0.6)
rect.hclust(as.hclust(res), k=6, border="red") # Clusteranzahl k anpassen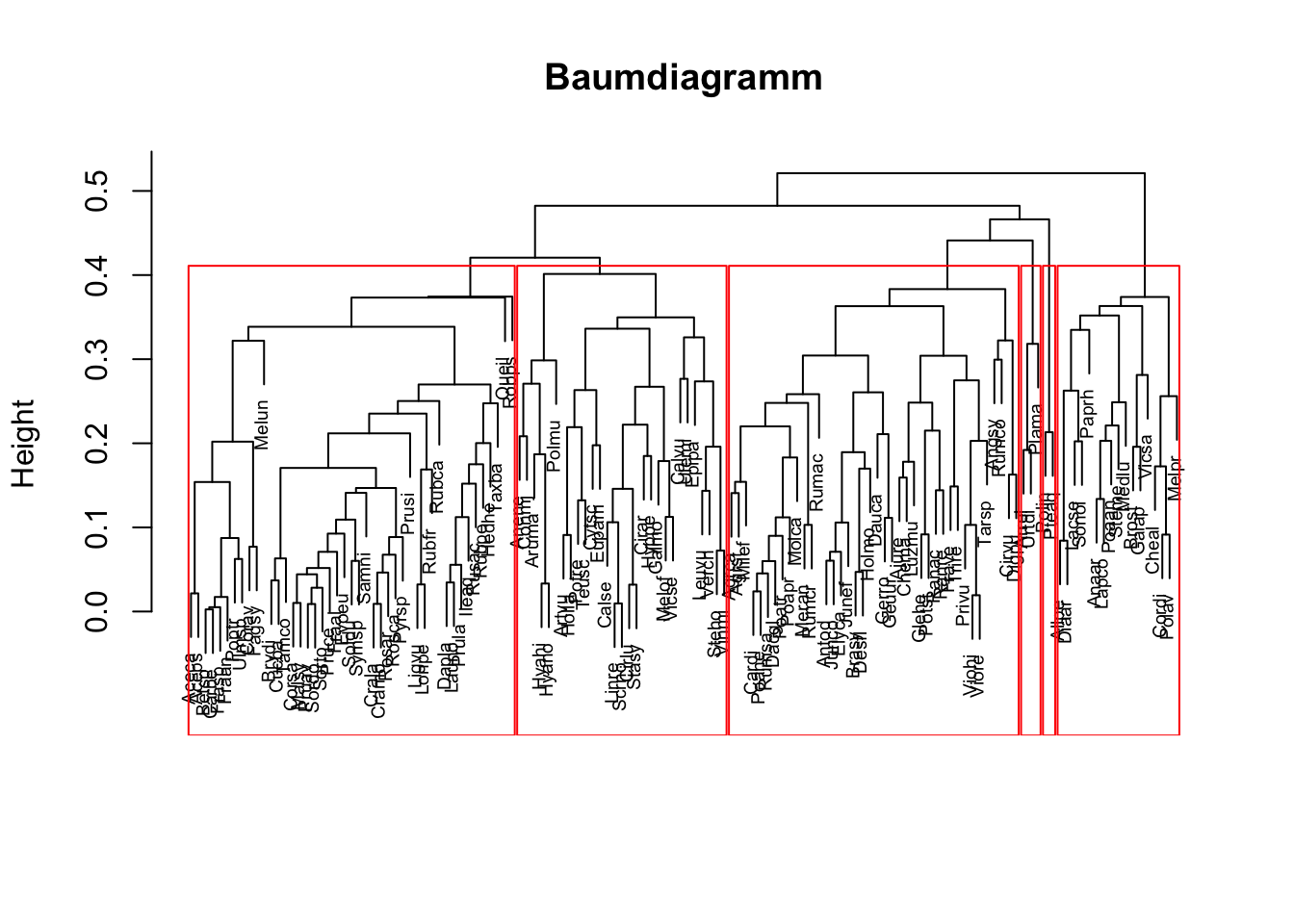
Hinweis 3: Vergleich zweier Clusterlösungen
# Beispiel: Vergleich von agglomerativer Clusteranalyse "average" und "Partitioning Around Medoids"
# für eine 6-Clusterlösung
library(fpc)
res1 <- agnes(d, method="average") # agglomerative Clusteranalyse, "average"
res2 <- pam(d, k=6) # Partitioning Around Medoids, Anzahl Cluster eingeben
cl.st <- cluster.stats(d, cutree(as.hclust(res1), k=6), res2$clustering) # d und Clusterzugehörigkeit
# Alle Statistiken mit cl.st. Interpretation vgl. ?cluster.stats
paste("Korrigierter Rand Index =", round(cl.st$corrected.rand,4))## [1] "Korrigierter Rand Index = 0.3999"Hierarchisch divisive Clusteranalyse
Datensatz einlesen, Variablen spezifizieren, Distanzmatrix berechnen
# Dateneingabe
data(plantTraits) # Mit ?plantTraits erhalten Sie die Variablenbeschreibungen
data <- plantTraits # Lassen Sie sich mit str(data) die Variablenklassen anzeigen.
# Zu verwendende Variablen
Variablen <- 1:31 # Spaltennummern der zu berücksichtigenden Variablen
Namen <- NULL # Spaltennummer mit den Zeilennamen. Verwenden der vorhandenen Zeilennamen: Namen <- NULL
# Distanzmatrix berechnen
# Wenn alle Variablen dasselbe Skalenniveau haben, kann unter "metric" zwischen "euclidean", manhattan"
# oder "gover" gewählt werden.
# Wenn die Variablen unterschiedliche Skalenniveaus aufweisen, kann man diese unter "type" spezifizieren
# ("ordratio"=ordinal, "asymm"=asymmetrisch binär und "symm" symmetrisch binär). Intervallskalierte und
# nominale Variablen werden automatisch erkannt und müssen nicht deklariert werden.
# Beispiel: d <- daisy(data2, type=list(ordratio = 3:5, symm = 9:10, asymm = 11:12))
# Diese Spaltennummern beziehen sich auf data2. Die Spalten 6:7 könnten intervallskalierte Variablen und
# die Spalte 8 eine nominale Variable mit 4 Stufen enthalten.
library(cluster); library(ade4)
if (!is.null(Namen)) rownames(data) <- data[,Namen] # Fallnamen als Zeilennamen definieren
data2 <- data[,Variablen, drop=FALSE]
data2 <- data2[rowSums(is.na(data2))!=ncol(data2), ] # Fälle ausschliessen, bei denen alle Werte fehlen
d <- daisy(data2, type=list(ordratio=4:11, symm=12:13, asymm=14:31)) # Spaltennummern von Data2!Clusteranalyse
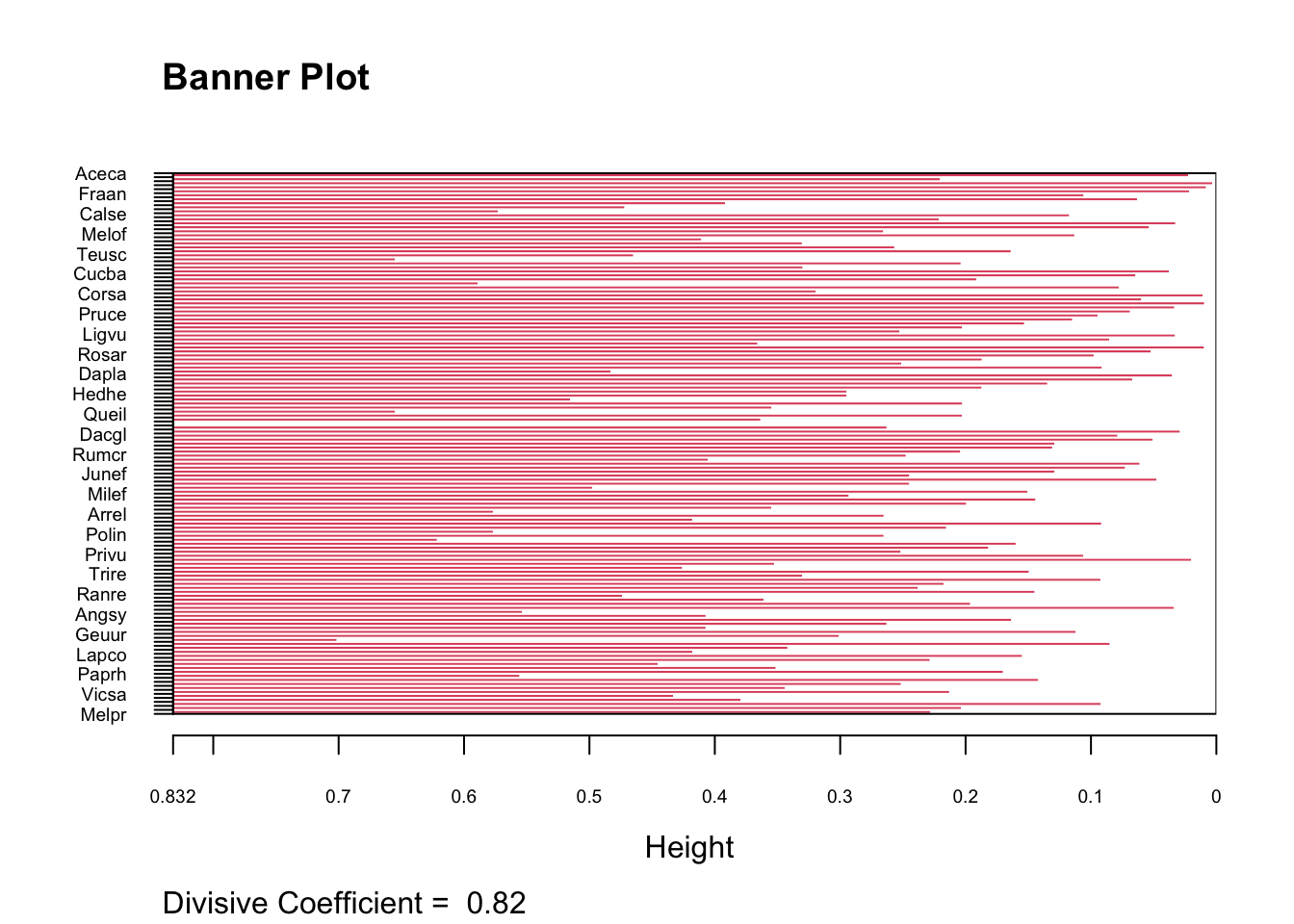
res <- diana(d, diss=TRUE, metric="euclidean")
res## Merge:
## [,1] [,2]
## [1,] -14 -20
## [2,] 1 -48
## [3,] -93 -117
## [4,] -31 -32
## [5,] -30 -72
## [6,] -135 -136
## [7,] 2 -47
## [8,] -1 -2
## [9,] -21 -35
## [10,] -25 -119
## [11,] -68 -70
## [12,] 3 -118
## [13,] -58 -59
## [14,] -36 -66
## [15,] -17 -33
## [16,] -15 -38
## [17,] -83 -111
## [18,] 4 -103
## [19,] 10 -69
## [20,] 5 12
## [21,] -10 -62
## [22,] -89 -129
## [23,] 15 -123
## [24,] 14 -95
## [25,] 20 -94
## [26,] 21 -41
## [27,] -28 -45
## [28,] 9 17
## [29,] -6 -39
## [30,] 11 -122
## [31,] -104 -105
## [32,] -12 -56
## [33,] -29 -86
## [34,] -54 -91
## [35,] 25 -46
## [36,] 18 -98
## [37,] 7 22
## [38,] -92 6
## [39,] -52 -53
## [40,] -74 -133
## [41,] 35 -44
## [42,] -18 -114
## [43,] 28 -85
## [44,] 26 -63
## [45,] 43 -84
## [46,] 24 -61
## [47,] -7 -121
## [48,] -67 -131
## [49,] -100 -101
## [50,] -49 -127
## [51,] -4 -78
## [52,] 41 -115
## [53,] -65 -116
## [54,] -5 -23
## [55,] -26 -40
## [56,] -60 -126
## [57,] -77 -80
## [58,] 54 -71
## [59,] 36 -96
## [60,] 46 -112
## [61,] 23 -107
## [62,] -27 13
## [63,] 48 -120
## [64,] -34 -128
## [65,] 52 -113
## [66,] -99 -106
## [67,] 33 -51
## [68,] -13 -88
## [69,] 45 -110
## [70,] -50 -132
## [71,] 32 -90
## [72,] 34 -134
## [73,] 8 37
## [74,] 42 19
## [75,] 67 -75
## [76,] 53 -82
## [77,] 72 49
## [78,] 16 -57
## [79,] 44 78
## [80,] 69 -79
## [81,] 59 31
## [82,] 47 -73
## [83,] 58 38
## [84,] 65 30
## [85,] -43 56
## [86,] -3 80
## [87,] 55 -37
## [88,] -11 -81
## [89,] -87 -108
## [90,] 74 40
## [91,] 51 63
## [92,] -55 -125
## [93,] 60 92
## [94,] 39 -109
## [95,] 27 84
## [96,] 68 61
## [97,] 50 77
## [98,] -24 85
## [99,] 29 -64
## [100,] 82 70
## [101,] -16 57
## [102,] 83 -124
## [103,] 64 -102
## [104,] 91 -42
## [105,] -8 62
## [106,] 66 -130
## [107,] 95 81
## [108,] -22 75
## [109,] 73 -19
## [110,] 86 79
## [111,] 87 94
## [112,] -9 111
## [113,] 90 98
## [114,] 99 76
## [115,] 88 71
## [116,] 102 97
## [117,] 100 108
## [118,] 114 101
## [119,] 113 -97
## [120,] 109 -76
## [121,] 116 105
## [122,] 107 93
## [123,] 110 104
## [124,] 122 103
## [125,] 121 112
## [126,] 118 117
## [127,] 120 119
## [128,] 115 89
## [129,] 123 128
## [130,] 96 124
## [131,] 129 125
## [132,] 130 106
## [133,] 127 132
## [134,] 131 126
## [135,] 133 134
## Order of objects:
## [1] Aceca Aceps Betsp Carbe Frasp Fraan Poptr Ulmsp Calvu Melun Calse Scrno
## [13] Cirlu Stasy Linre Melof Vicse Cirar Eupam Hyppe Teusc Pteaq Aruma Polmu
## [25] Brydi Cucba Tamco Rubpe Corav Fagsy Corsa Malsy Pruav Sordo Sorto Pruce
## [37] Fraal Evoeu Soldu Samni Ligvu Lonpe Symsp Crala Cramo Rosar Pyrsp Prusi
## [49] Rosca Rubca Dapla Launo Prula Ileaq Rusac Hedhe Taxba Cytsc Ulemi Robps
## [61] Queil Rubfr Urtdi Agrca Cardi Dacgl Poane Rumsa Poatr Poapr Rumcr Molca
## [73] Antod Junco Elyca Junef Brasy Desfl Holmo Agrst Milef Leuvu Verch Steho
## [85] Epipa Arrel Plama Artvu Holla Potre Polin Rumac Ajure Chema Luzmu Privu
## [97] Viohi Viore Tarsp Frave Trire Glehe Potst Vinmi Ranac Ranre Anene Conmj
## [109] Hyahi Hyano Angsy Cirvu Digpu Dauca Gerro Geuur Rumco Allpe Diaar Lacse
## [121] Lapco Sonol Poaan Brost Meran Paprh Anaar Steme Medlu Galap Vicsa Cheal
## [133] Cordi Polav Galmo Melpr
## Height:
## [1] 0.021588653 0.219866071 0.002521339 0.007449479 0.020943426 0.105134680
## [7] 0.062500000 0.391118516 0.471304055 0.572275354 0.116588838 0.220456431
## [13] 0.031947442 0.053238428 0.264898990 0.112476809 0.410028860 0.329768273
## [19] 0.256086492 0.163216214 0.464285714 0.654383628 0.203116867 0.329241071
## [25] 0.036989796 0.063775510 0.190584416 0.588293651 0.077081144 0.318646409
## [31] 0.010204082 0.059126984 0.008928571 0.032738095 0.068091631 0.093975469
## [37] 0.114135864 0.152501910 0.202030812 0.251956377 0.032196970 0.084706960
## [43] 0.365259740 0.009091368 0.051547238 0.097069597 0.186286798 0.250574946
## [49] 0.090604708 0.482447240 0.034554731 0.066326531 0.134044527 0.186456401
## [55] 0.294155844 0.294155844 0.514705882 0.201937316 0.354087089 0.654383628
## [61] 0.202155483 0.362857264 0.832028266 0.262223053 0.028290349 0.078070084
## [67] 0.050045271 0.128323118 0.130145651 0.203482937 0.246911674 0.404762657
## [73] 0.060455370 0.072065329 0.128332043 0.244118755 0.047068228 0.244118755
## [79] 0.496947415 0.149941554 0.292443986 0.143524814 0.198700373 0.354229267
## [85] 0.576214205 0.264572201 0.417248764 0.091005176 0.214713968 0.576214205
## [91] 0.264610390 0.620861637 0.159272765 0.181060606 0.251116071 0.105385897
## [97] 0.019382346 0.351996859 0.425296381 0.148709537 0.329527417 0.091695624
## [103] 0.216643476 0.237247073 0.144163546 0.473063973 0.360321970 0.195549975
## [109] 0.033279221 0.552849928 0.406560562 0.162796323 0.262277670 0.406560562
## [115] 0.111391673 0.300453694 0.700843644 0.084271035 0.341303664 0.417230828
## [121] 0.154213797 0.227782170 0.444786517 0.350708039 0.169398726 0.554996393
## [127] 0.141409529 0.250806903 0.343435333 0.212269470 0.432431563 0.378573683
## [133] 0.091471029 0.202838828 0.227323458
## Divisive coefficient:
## [1] 0.8228246
##
## Available components:
## [1] "order" "height" "dc" "merge" "diss" "call"
## [7] "order.lab"Baumdiagramm für eher kleine Fallzahlen
plot(res, which.plots=2, main="Baumdiagramm", xlab="", sub="", cex=0.6)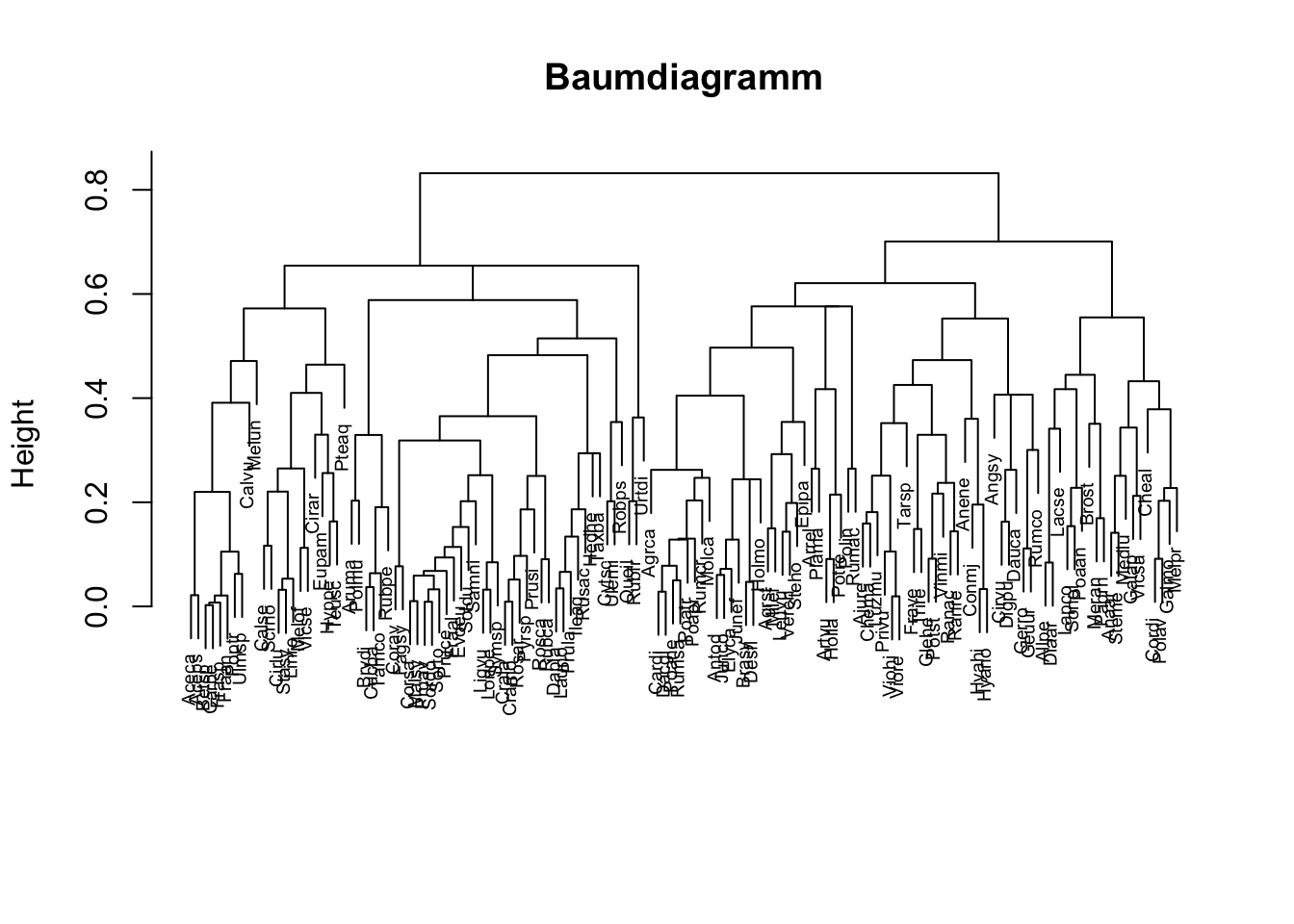
Scree Plot für hohe Fallzahlen
# Es wird das Kriterium "height" in Abhängigkeit der letzten 10 Fusionierungsschritte dargestellt.
height <- sort(res$height)
Schritt <- (length(height)-9):length(height)
height <- height[(length(height)-9):length(height)]
data4 <- data.frame(Schritt, height)
library(ggplot2)
ggplot(data4, aes(x=Schritt, y=height)) + geom_line() +
scale_x_continuous(breaks=Schritt)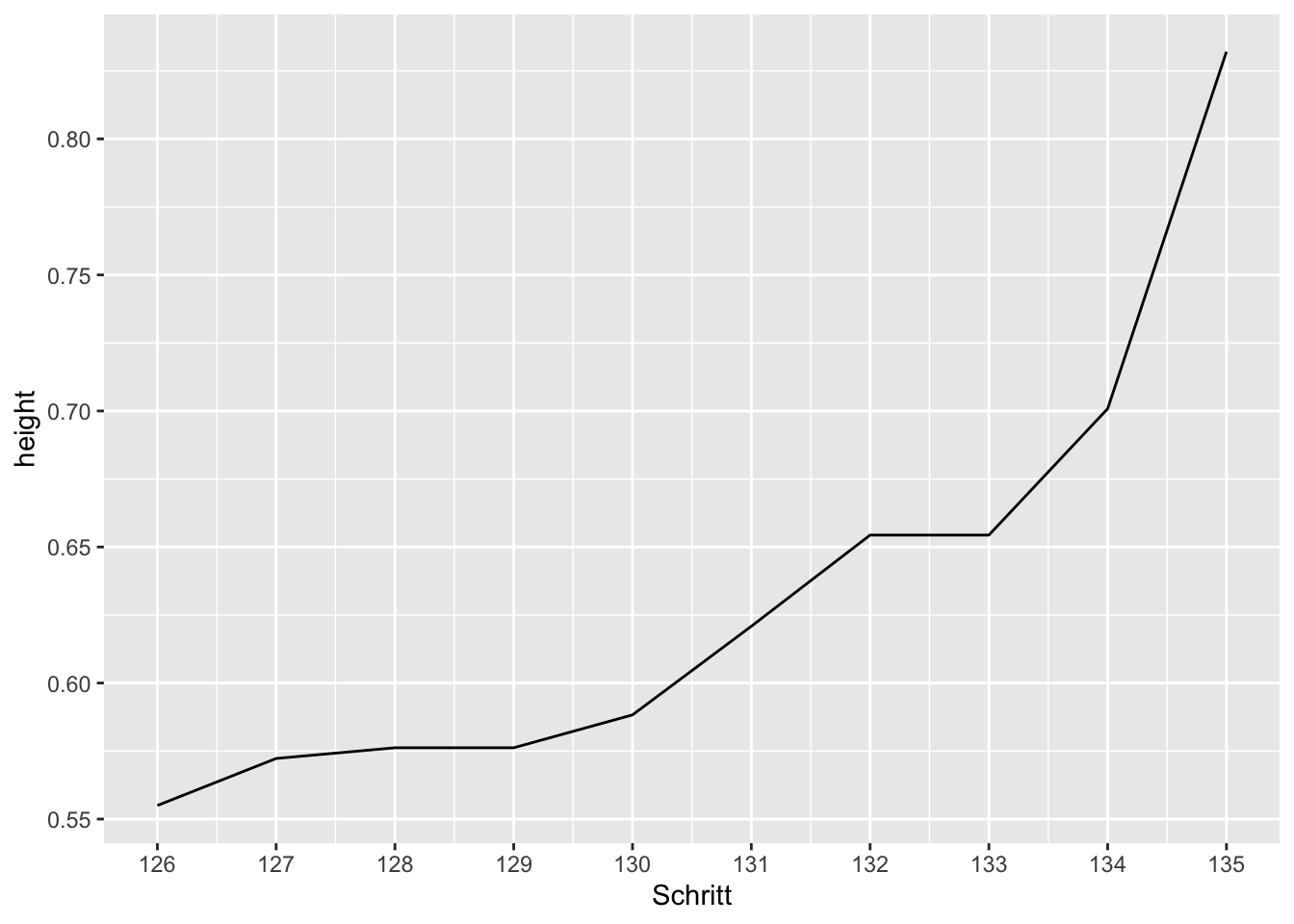
Hinweis 1: Variable mit der Clusterzugehörigkeit ins Datenfile einfügen
# Vektor "Cluster" mit Clusterzugehörigkeit
# Cluster <- cutree(as.hclust(res), k = 6) # Clusteranzahl k anpassen
# Vektor "Cluster" ins Datenfile "data" einfügen
# dat <- numeric(length=nrow(data)); dat[dat==0] <- NA
# dat[rownames(data) %in% names(Cluster)] <- Cluster
# data$Cluster <- dat
Hinweis 2: Baumdiagramm mit markierten Clustern für eine vorgegebene Clusteranzahl k
plot(res, which.plots=2, main="Baumdiagramm", xlab="", sub="", cex=0.6)
rect.hclust(as.hclust(res), k=6, border="red") # Clusteranzahl k anpassen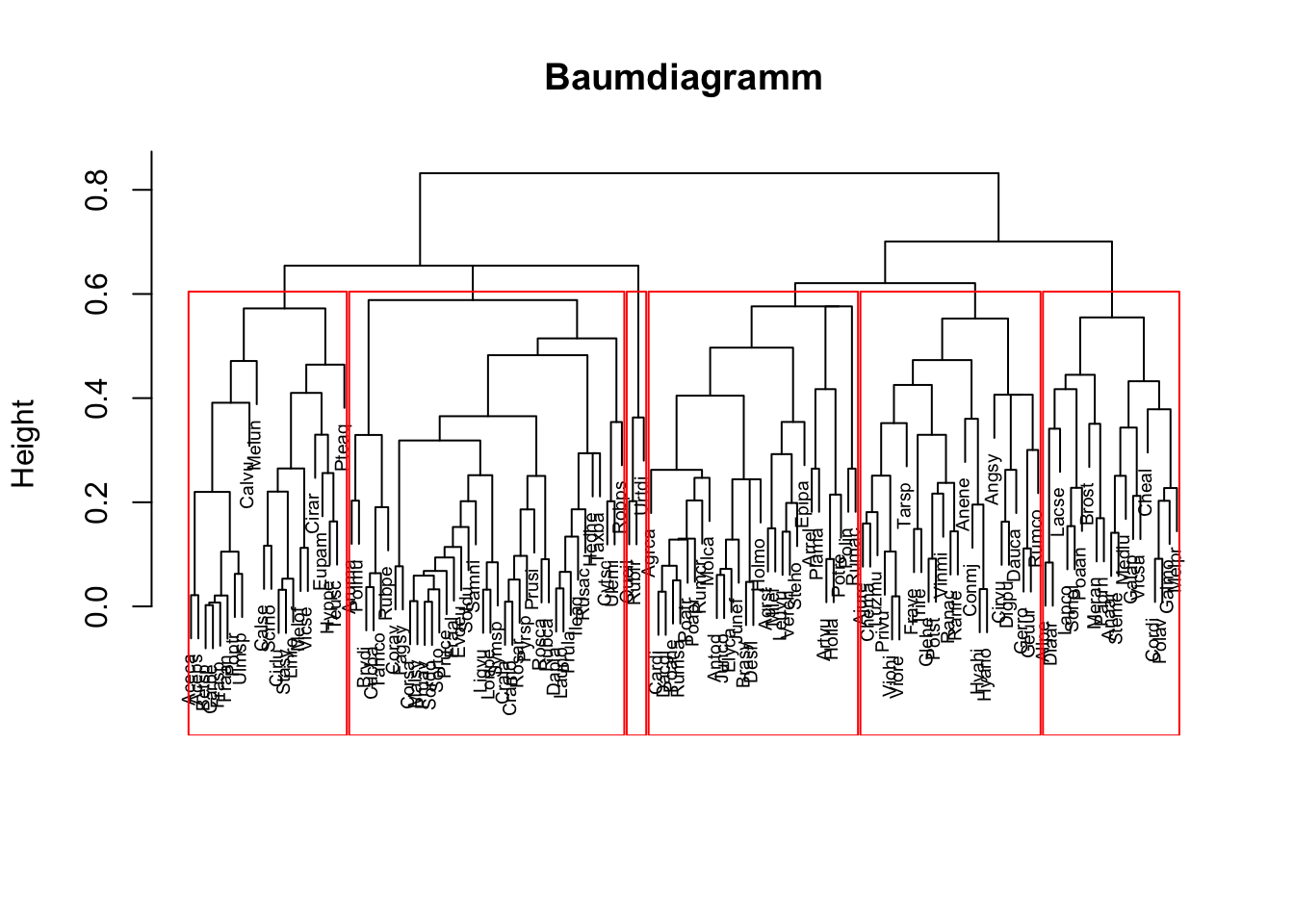
Hinweis 3: Vergleich zweier Clusterlösungen
# Beispiel: Vergleich von agglomerativer und divisiver Clusteranalyse für eine 6-Clusterlösung
library(fpc)
res1 <- agnes(d, method="average") # hierarchisch agglomerative Clusteranalyse
res2 <- diana(d, diss=TRUE, metric="euclidean") # hierarchisch divisive Clusteranalyse
cl.st <- cluster.stats(d, cutree(as.hclust(res1), k=6), cutree(as.hclust(res2), k=6))
# Alle Statistiken mit cl.st. Interpretation vgl. ?cluster.stats
paste("Korrigierter Rand Index =", round(cl.st$corrected.rand,4))## [1] "Korrigierter Rand Index = 0.4612"Partitionierende Clusteranalyse
Datensatz einlesen, Variablen und Anzahl Cluster spezifizieren, Distanzmatrix berechnen
# Dateneingabe
data(plantTraits) # Mit ?plantTraits erhalten Sie die Variablenbeschreibungen
data <- plantTraits # Lassen Sie sich mit str(data) die Variablenklassen anzeigen.
# Zu verwendende Variablen
Variablen <- 1:31 # Spaltennummern der zu berücksichtigenden Variablen
Namen <- NULL # Spaltennummer mit den Zeilennamen. Verwenden der vorhandenen Zeilennamen: Namen <- NULL
k <- 6 # Anzahl Cluster eingeben
# Distanzmatrix berechnen
# Wenn alle Variablen dasselbe Skalenniveau haben, kann unter "metric" zwischen "euclidean", manhattan"
# oder "gover" gewählt werden.
# Wenn die Variablen unterschiedliche Skalenniveaus aufweisen, kann man diese unter "type" spezifizieren
# ("ordratio"=ordinal, "asymm"=asymmetrisch binär und "symm" symmetrisch binär). Intervallskalierte und
# nominale Variablen werden automatisch erkannt und müssen nicht deklariert werden.
# Beispiel: d <- daisy(data2, type=list(ordratio = 3:5, symm = 9:10, asymm = 11:12))
# Diese Spaltennummern beziehen sich auf data2. Die Spalten 6:7 könnten intervallskalierte Variablen und
# die Spalte 8 eine nominale Variable mit 4 Stufen enthalten.
library(cluster); library(ade4)
if (!is.null(Namen)) rownames(data) <- data[,Namen] # Fallnamen als Zeilennamen definieren
data2 <- data[, Variablen, drop=FALSE]
data2 <- data2[rowSums(is.na(data2))!=ncol(data2), ] # Fälle ausschliessen, bei denen alle Werte fehlen
d <- daisy(data2, type=list(ordratio=4:11, symm=12:13, asymm=14:31)) # Spaltennummern von Data2!Abschätzen der optimalen Clusteranzahl
# pam
if (attr(d,"Size")<20) asw <- numeric(attr(d,"Size")-1) else asw <- numeric(20)
for (i in 2:length(asw)) asw[i] <- pam(d, i)$silinfo$avg.width
k.best <- which.max(asw)
plot(1:length(asw), asw, type= "h", main = "pam() clustering assessment",
xlab= "k (# clusters)", ylab = "average silhouette width")
axis(1, k.best, paste("best",k.best,sep="\n"), col = "red", col.axis = "red")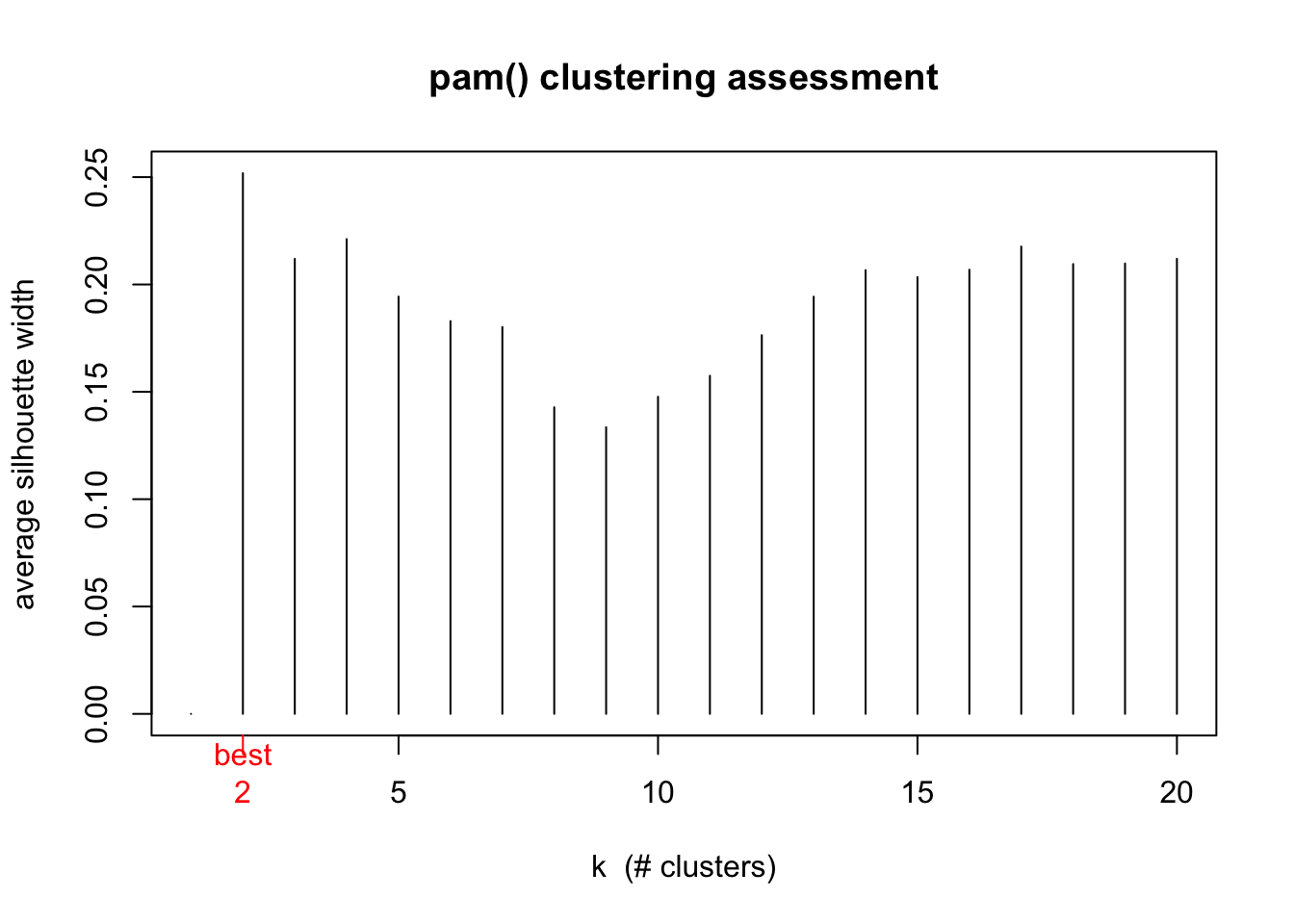
# Variante für K-Means (für dieses Besipiel wegen der unterschiedlichen Skalenniveaus nicht sinnvoll)
# if (attr(d,"Size")<20) asw <- numeric(attr(d,"Size")-1) else asw <- 20
# wss <- (nrow(data2)-1)*sum(apply(data2,2,var))
# for (i in 2:asw) wss[i] <- sum(kmeans(data2, centers=i)$withinss)
# plot(1:asw, wss, type="b", xlab="Number of Clusters", ylab="Within groups sum of squares"Clusteranalyse: Partitioning Around Medoids und K-Means
Wir verwenden die Funktion pam() für Partitioning Around Medoids des Pakets cluster, da sie den Datensatz oder die Distanzmatrix als Argument zulässt.
Wenn Sie einen Datensatz mit intervallskalierten Daten haben, können Sie mit der Funktion kmeans() eine K-Means Clusteranalyse rechnen und den Datensatz data2 ohne fehlende Werte als erstes Argument eingeben. Im Beispiel ist der Code für diese Alternative jeweils angegeben. Bei unterschiedlichen Varianzen sollten die Variablen zuerst standardisiert werden: scale(data2).
# pam
res <- pam(d, k=k); res## Medoids:
## ID
## [1,] "48" "Frasp"
## [2,] "21" "Cardi"
## [3,] "62" "Junco"
## [4,] "39" "Diaar"
## [5,] "69" "Linre"
## [6,] "103" "Rosar"
## Clustering vector:
## Aceca Aceps Agrca Agrst Ajure Allpe Anaar Anene Angsy Antod Arrel Artvu Aruma
## 1 1 2 2 3 4 4 5 4 3 3 2 5
## Betsp Brasy Brost Brydi Calse Calvu Carbe Cardi Cheal Chema Cirar Cirlu Cirvu
## 1 3 4 5 5 6 1 2 5 3 5 5 4
## Conmj Corav Cordi Corsa Crala Cramo Cucba Cytsc Dacgl Dapla Dauca Desfl Diaar
## 2 1 4 6 6 6 5 6 2 6 4 3 4
## Digpu Elyca Epipa Eupam Evoeu Fagsy Fraal Fraan Frasp Frave Galap Galmo Gerro
## 4 3 3 5 6 1 6 1 1 3 4 5 4
## Geuur Glehe Hedhe Holla Holmo Hyahi Hyano Hyppe Ileaq Junco Junef Lacse Lapco
## 3 2 6 3 3 2 2 5 6 3 3 4 4
## Launo Leuvu Ligvu Linre Lonpe Luzmu Malsy Medlu Melof Melpr Melun Meran Milef
## 6 5 6 5 6 2 6 4 5 4 5 2 2
## Molca Paprh Plama Poaan Poane Poapr Poatr Polav Polin Polmu Poptr Potre Potst
## 2 4 3 4 2 2 2 4 5 5 1 5 2
## Privu Pruav Pruce Prula Prusi Pteaq Pyrsp Queil Ranac Ranre Robps Rosar Rosca
## 3 6 5 6 6 5 6 6 3 3 5 6 6
## Rubca Rubfr Rubpe Rumac Rumco Rumcr Rumsa Rusac Samni Scrno Soldu Sonol Sordo
## 6 6 6 2 3 2 2 6 6 5 5 4 6
## Sorto Stasy Steho Steme Symsp Tamco Tarsp Taxba Teusc Trire Ulemi Ulmsp Urtdi
## 6 5 2 4 6 5 3 1 5 3 6 1 3
## Verch Vicsa Vicse Vinmi Viohi Viore
## 5 4 5 3 3 3
## Objective function:
## build swap
## 0.1834797 0.1794031
##
## Available components:
## [1] "medoids" "id.med" "clustering" "objective" "isolation"
## [6] "clusinfo" "silinfo" "diss" "call"# Variante für K-Means
# res <- kmeans(na.omit(data2), k, nstart = 100); resSilhouette Plot für PAM
plot(res, main="Silhouette Plot")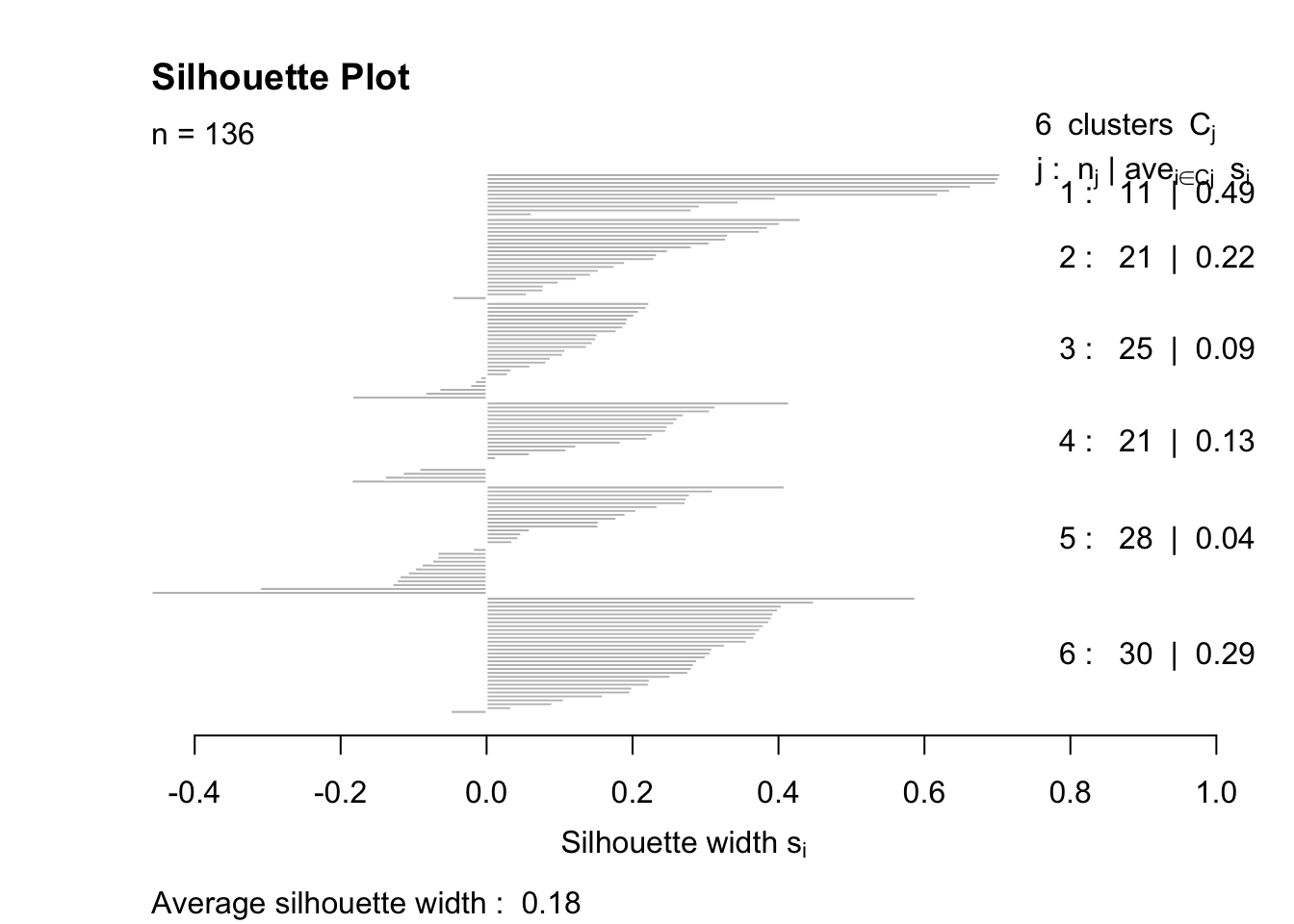
Überprüfung der Stabilität mit Bootstrap
k = 6 # Anzahl Cluster eingeben
library(fpc)
stab <- clusterboot(d, B=100, distances=TRUE, bootmethod="boot", clustermethod=pamkCBI, k=k, showplots=FALSE)stab # Interpretation vgl. ?clusterboot## * Cluster stability assessment *
## Cluster method: pam/estimated k
## Full clustering results are given as parameter result
## of the clusterboot object, which also provides further statistics
## of the resampling results.
## Number of resampling runs: 100
##
## Number of clusters found in data: 6
##
## Clusterwise Jaccard bootstrap (omitting multiple points) mean:
## [1] 0.7182704 0.5219917 0.5182478 0.7498535 0.5999396 0.7482602
## dissolved:
## [1] 27 49 59 14 35 5
## recovered:
## [1] 63 14 14 52 33 47# Variante für kmeans (für dieses Besipiel wegen der unterschiedlichen Skalenniveaus nicht sinnvoll)
# k = 6 # Anzahl Cluster eingeben
# stab <- clusterboot(na.omit(data2), B=100, distances=FALSE, bootmethod="boot", clustermethod=kmeansCBI, k=k, showplots=FALSE)
# stab
Hinweis 1: Vektor mit der Clusterzugehörigkeit ins Datenfile einfügen
# Vektor "Cluster" mit Clusterzugehörigkeit
# Cluster <- res$clustering # Alternative für pam
# Cluster <- res$cluster # Alternative für K-Means
# Vektor "Cluster" ins Datenfile "data" einfügen
# dat <- numeric(length=nrow(data)); dat[dat==0] <- NA
# dat[rownames(data) %in% names(Cluster)] <- Cluster
# data$Cluster <- dat
Hinweis 2: Vergleich zweier Clusterlösungen
# Beispiel: Vergleich von agglomerativer Clusteranalyse und Partitioning Around Medoids
# für eine 6-Clusterlösung
library(fpc)
res1 <- hclust(d, method="ward.D2") # agglomerative Clusteranalyse, "ward.D2"
res2 <- pam(d, k=6) # Partitioning Around Medoids, Anzahl Cluster eingeben
cl.st <- cluster.stats(d, cutree(res1, k=6), res2$clustering) # d und Clusterzugehörigkeit
# Alle Statistiken mit cl.st. Interpretation vgl. ?cluster.stats
paste("Korrigierter Rand Index =", round(cl.st$corrected.rand,4))## [1] "Korrigierter Rand Index = 0.4879"