Multiple lineare Regression
Ausgehend von den Rohdaten oder der Korrelationsmatrix
Rohdaten
Erforderliche Pakete laden
library(leaps) # Modellvereinfachung
library(psych) # Grafik
library(olsrr) # Ergebnisse, GrafikDatensatz einlesen und Modell spezifizieren
# Dateneingabe
data <- read.table(file="https://mmi.psycho.unibas.ch/r-toolbox/data/Partial.txt", header=TRUE)
head(data) # zeigt die ersten 6 Zeilen des Datensatzes## CASE TIME PUBS CITS SALARY FEMALE
## 1 1 3 18 50 51876 1
## 2 2 6 3 26 54511 1
## 3 3 3 2 50 53425 1
## 4 4 8 17 34 61863 0
## 5 5 9 11 41 52926 1
## 6 6 6 6 37 47034 0# Regressionsmodell spezifizieren
model <- SALARY~TIME+PUBS+CITS+FEMALESignifikanztest
# Überprüfen des Gesamtmodells (Signifikanztests, Regressionskoeffizienten)
variablen <- all.vars(model)
data2 <- na.omit(data[, variablen, drop=FALSE])
fit <- lm(model, data2)
# R2, Test für Gesamtmodell, Regressionskoeffizienten
library(olsrr)
res1 <- ols_regress(model, data2)
if (length(all.vars(model))>2) {
# Bivariate Korrelationen, Partial- und Semipartialkorrelationen
res2 <- ols_correlations(fit)
list("R2, Test für das Gesamtmodell und Regressionskoeffizienten"=res1, "Bivariate Korrelationen, Partial- und Semipartialkorrelationen"=res2)
} else {
list("R2, Test für das Gesamtmodell und Regressionskoeffizienten"=res1)
}## $`R2, Test für das Gesamtmodell und Regressionskoeffizienten`
## Model Summary
## --------------------------------------------------------------------
## R 0.709 RMSE 7076.846
## R-Squared 0.503 Coef. Var 12.910
## Adj. R-Squared 0.468 MSE 50081752.352
## Pred R-Squared 0.400 MAE 5364.108
## --------------------------------------------------------------------
## RMSE: Root Mean Square Error
## MSE: Mean Square Error
## MAE: Mean Absolute Error
##
## ANOVA
## ----------------------------------------------------------------------------
## Sum of
## Squares DF Mean Square F Sig.
## ----------------------------------------------------------------------------
## Regression 2891959939.332 4 722989984.833 14.436 0.0000
## Residual 2854659884.039 57 50081752.352
## Total 5746619823.371 61
## ----------------------------------------------------------------------------
##
## Parameter Estimates
## -------------------------------------------------------------------------------------------------
## model Beta Std. Error Std. Beta t Sig lower upper
## -------------------------------------------------------------------------------------------------
## (Intercept) 39587.350 2717.480 14.568 0.000 34145.690 45029.009
## TIME 857.006 287.946 0.378 2.976 0.004 280.404 1433.608
## PUBS 92.746 85.928 0.134 1.079 0.285 -79.322 264.815
## CITS 201.931 57.509 0.357 3.511 0.001 86.771 317.091
## FEMALE -917.767 1859.936 -0.047 -0.493 0.624 -4642.225 2806.690
## -------------------------------------------------------------------------------------------------
##
## $`Bivariate Korrelationen, Partial- und Semipartialkorrelationen`
## Correlations
## -------------------------------------------
## Variable Zero Order Partial Part
## -------------------------------------------
## TIME 0.608 0.367 0.278
## PUBS 0.506 0.142 0.101
## CITS 0.550 0.422 0.328
## FEMALE -0.201 -0.065 -0.046
## -------------------------------------------Kollinearitätsdiagnostik
library(olsrr)
ols_coll_diag(fit)## Tolerance and Variance Inflation Factor
## ---------------------------------------
## Variables Tolerance VIF
## 1 TIME 0.5411117 1.848047
## 2 PUBS 0.5669776 1.763738
## 3 CITS 0.8418826 1.187814
## 4 FEMALE 0.9498246 1.052826
##
##
## Eigenvalue and Condition Index
## ------------------------------
## Eigenvalue Condition Index intercept TIME PUBS CITS
## 1 3.95178690 1.000000 0.0064170506 0.008406453 0.01084481 0.007340295
## 2 0.65878913 2.449196 0.0007861334 0.020699491 0.03623289 0.001648078
## 3 0.21340107 4.303269 0.0971898337 0.025687174 0.38746940 0.179662771
## 4 0.10611153 6.102608 0.0042569904 0.926445674 0.55482486 0.078365963
## 5 0.06991137 7.518355 0.8913499919 0.018761207 0.01062804 0.732982893
## FEMALE
## 1 0.01599588
## 2 0.63716396
## 3 0.19897663
## 4 0.01168851
## 5 0.13617503Diagnostikplots
Matrix der Streudiagramme aller beteiligten Variablen
library(psych)
pairs.panels(data2)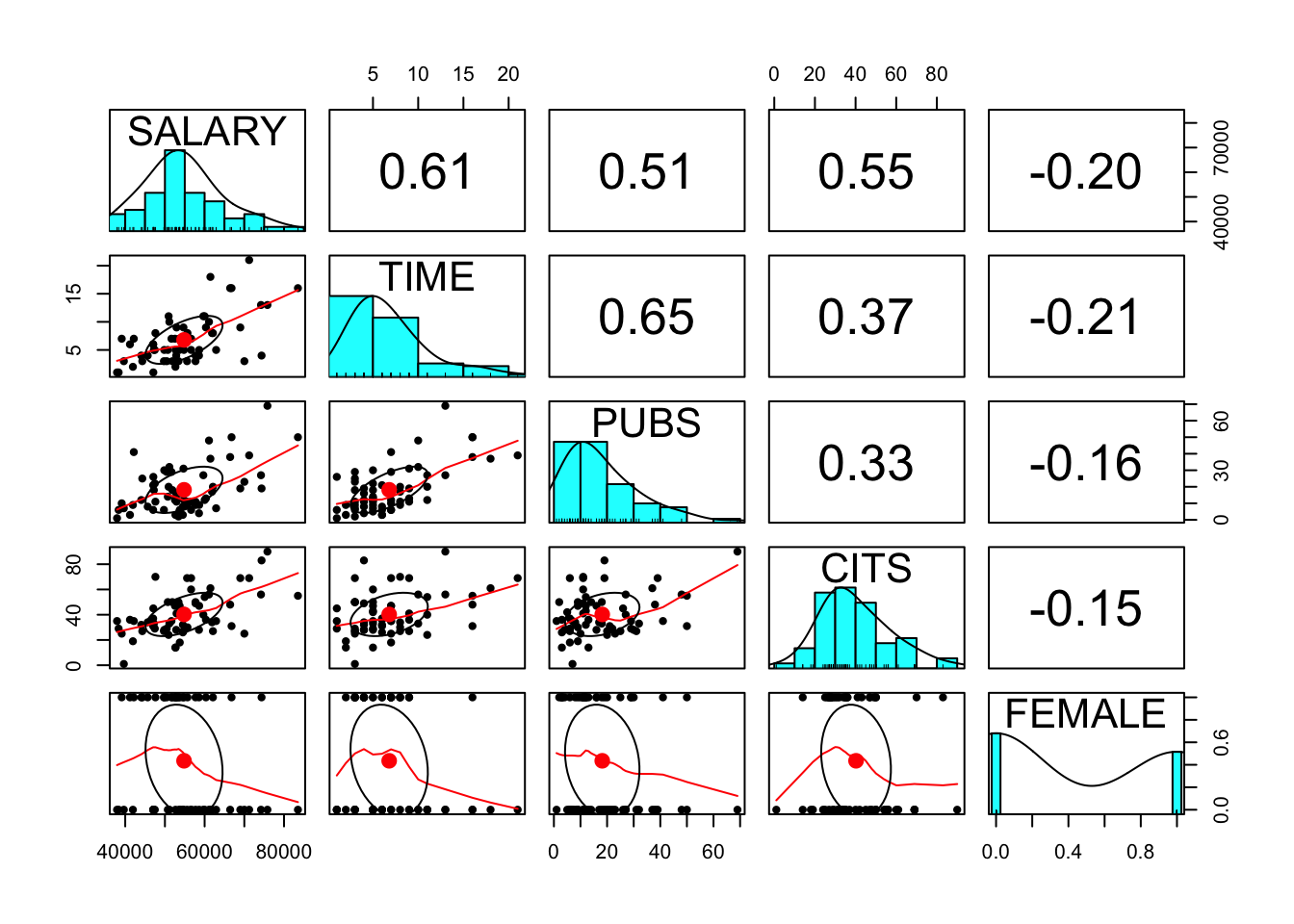
Residuals vs. Fitted, Q-Q-Plot der Residuen, Scale-Location, Residuals vs. Leverage + Cook’s Distance
par(mfrow=c(2,2), bty="n")
plot(fit)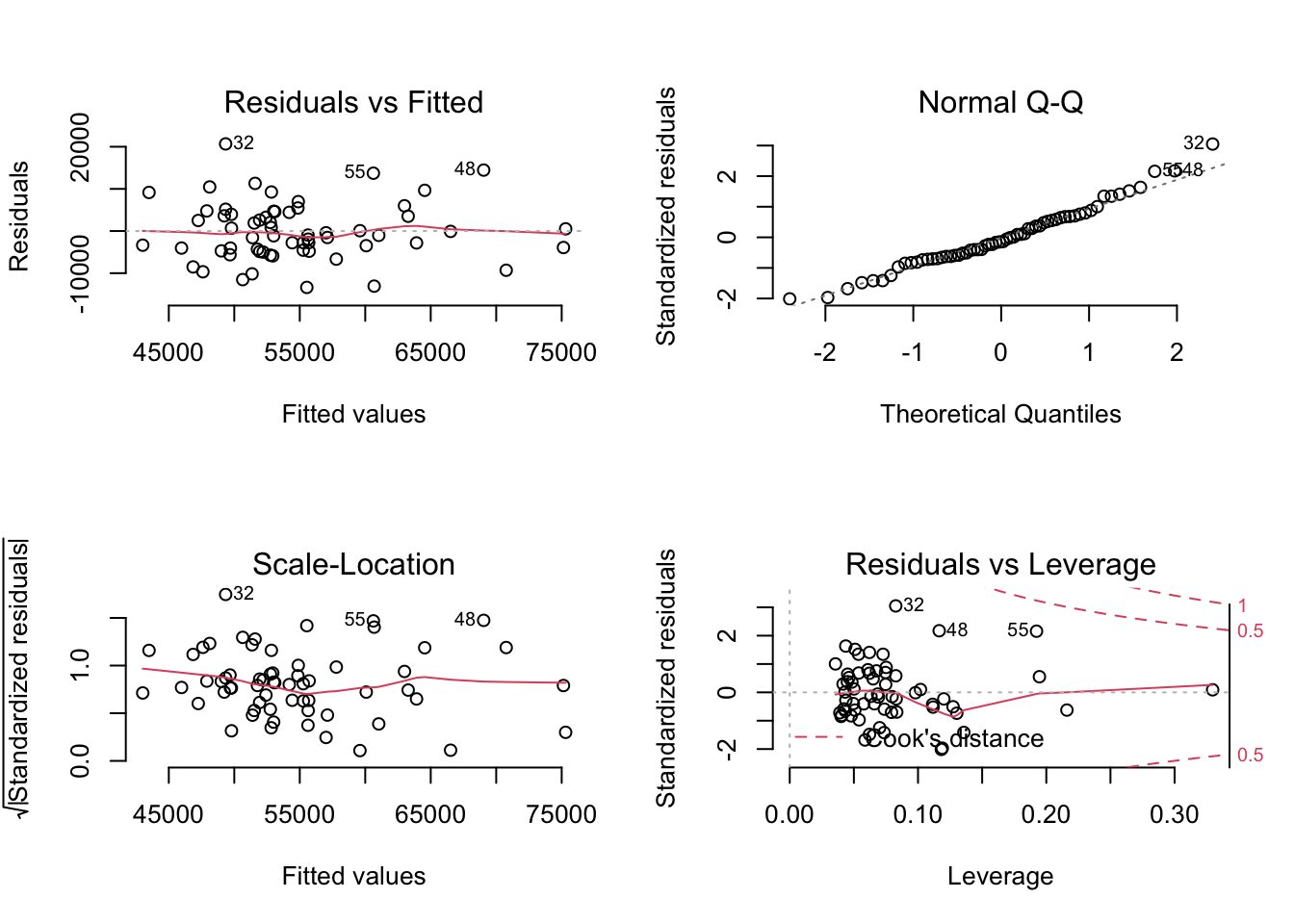
Weitere Diagnostikplots incl. Ausgabe der Zeilennummern kritischer Fälle vgl. https://cran.r-project.org/web/packages/olsrr/olsrr.pdf
Deleted Studenticed Residuals: DSR <- ols_srsd_plot(fit); DSR
Cook’s Distance: CD <- ols_cooksd_barplot(fit); CD
dfBeta(s): DB <- ols_dfbetas_panel(fit); DB
Studentized Residuals vs Leverage: SRL <- ols_rsdlev_plot(model); SRL
Residual Plus Component Plot: ols_rpc_plot(fit)
Modellvereinfachung
Vollständige Suche nach besten Modellen mit 1, 2, … Prädiktoren
library(leaps)
nvmax <- length(variablen)-1
a <- regsubsets(model, data2, nvmax=nvmax, nbest=1, method="exhaustive") # nvmax anpassen
# Erklärte Varianzanteile
R2 <- summary(a)$rsq
# Delta R-Quadrate
delta.R2 <- R2-c(0,R2[-nvmax])
# F-Werte
df1 <- rep(1,nvmax); df2 <- nrow(data)-2:(nvmax+1)
F.Wert <- delta.R2/(1-R2)*df2
p.Wert <- pf(F.Wert, df1, df2, lower.tail=FALSE)
list("Tabelle mit den besten Modellen"=summary(a), "Tabelle mit erklärten Varianzanteilen und delta-R2"=data.frame(R2,
delta.R2, df1, df2, F.Wert, p.Wert))## $`Tabelle mit den besten Modellen`
## Subset selection object
## Call: regsubsets.formula(model, data2, nvmax = nvmax, nbest = 1, method = "exhaustive")
## 4 Variables (and intercept)
## Forced in Forced out
## TIME FALSE FALSE
## PUBS FALSE FALSE
## CITS FALSE FALSE
## FEMALE FALSE FALSE
## 1 subsets of each size up to 4
## Selection Algorithm: exhaustive
## TIME PUBS CITS FEMALE
## 1 ( 1 ) "*" " " " " " "
## 2 ( 1 ) "*" " " "*" " "
## 3 ( 1 ) "*" "*" "*" " "
## 4 ( 1 ) "*" "*" "*" "*"
##
## $`Tabelle mit erklärten Varianzanteilen und delta-R2`
## R2 delta.R2 df1 df2 F.Wert p.Wert
## 1 0.3695369 0.369536855 1 60 35.1681324 1.606099e-07
## 2 0.4907768 0.121239925 1 59 14.0471905 4.078031e-04
## 3 0.5011234 0.010346656 1 58 1.2029149 2.772728e-01
## 4 0.5032454 0.002121955 1 57 0.2434832 6.235975e-01
Hinweis : Variable mit geschätzten y-Werten ins Datenfile einfügen
# vektor <- fit$fitted.values # Residuen: fit$residuals
# dat <- numeric(length=nrow(data)); dat[dat==0] <- NA
# dat[rownames(data) %in% names(vektor)] <- vektor
# data$fitted <- datGrafiken
Korrelationsmatrix
Erforderliche Pakete laden
library(psych) # RegressionsanalyseDatensatz einlesen und Modell spezifizieren
# Dateneingabe
# Korrelationsmatrix: Die obere Dreiecksmatrix darf Nullen enthalten.
data <- read.table(file="https://mmi.psycho.unibas.ch/r-toolbox/data/Partial.txt", header=TRUE)
data <- cor(data) # Korrelationsmatrix
y <- 5 # Spaltennummer der Zielvariable
x <- c(2:4, 6) # Spaltennumern der Prädiktoren
n <- 62 # StichprobengrösseSignifikanztest
data <- as.matrix(data)
data[upper.tri(data)] <- t(data)[upper.tri(data)]
rownames(data) <- colnames(data)
library(psych)
res <- setCor(y=y,x=x,data=data,n.obs=n)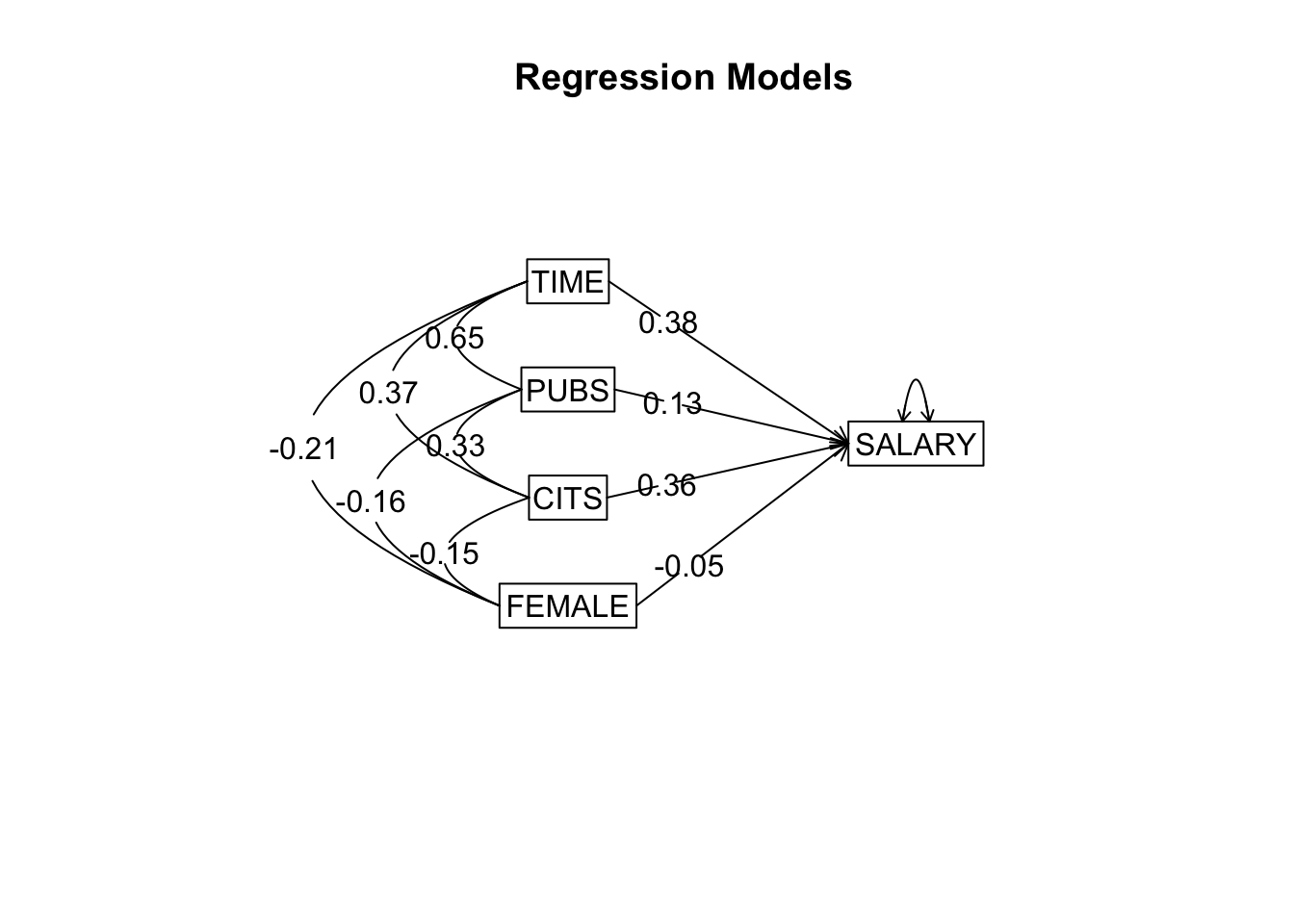
# Model Summary
res2 <- data.frame(res$R, res$R2, res$shrunkenR2)
names(res2) <- c("R", "R-Squared", "Adj. R-Squared"); rownames(res2)[1] <- ""
# ANOVA (Test für das Gesamtmodell)
res3 <- data.frame(res$R2, res$df[1], res$df[2], res$F, res$probF)
names(res3) <- c("R-Squared", "df1", "df2", "F", "Sig"); rownames(res3)[1] <- ""
# Parameter Estimates
res4 <- data.frame(res$coefficients, res$se, res$t, res$Probability)
names(res4) <- c("Std.Beta","Std.Error","t","Sig")
list("Model Summary"=res2, ANOVA=res3, "Parameter Estimates"=res4, Varianzinflationsfaktoren=res$VIF)## $`Model Summary`
## R R-Squared Adj. R-Squared
## 0.7093979 0.5032454 0.4683854
##
## $ANOVA
## R-Squared df1 df2 F Sig
## 0.5032454 4 57 14.4362 3.356914e-08
##
## $`Parameter Estimates`
## Std.Beta Std.Error t Sig
## TIME 0.37771405 0.12690832 2.9762750 0.0042758871
## PUBS 0.13381707 0.12397970 1.0793467 0.2849794117
## CITS 0.35725189 0.10174375 3.5112906 0.0008794694
## FEMALE -0.04726573 0.09578816 -0.4934402 0.6235974960
##
## $Varianzinflationsfaktoren
## TIME PUBS CITS FEMALE
## 1.848047 1.763738 1.187814 1.052826