Liniendiagramm für Häufigkeiten
ein- bis dreifaktorieller Designs
Erforderliche Pakete laden
library(ggplot2) # GrafikEinfaktoriell
Datensatz einlesen und Variable spezifizieren
# Datensatz 'data': pro Versuchsperson eine Zeile
# Häufigkeitstabellen zuerst umwandeln, vgl. Umwandlung von Datenstrukturen
Master <- rep(c("Entwicklung, Persoenlichkeit", "keine Vorstellung", "klinische Psychologie, Neuro", "Pflegewissenschaften", "Sozial, Wirtschaft, Entscheidung"), c(18,81,44,26,20))
data <- data.frame(Master)
# Variablen spezifizieren
Faktor <- "Master" # Name der Gruppenvariable eingebenLiniendiagramm
data2 <- na.omit(data[, Faktor, drop=FALSE]); library(ggplot2)
data.aggr <- data.frame(xtabs(paste("~", Faktor), data2))
# Schwarzweissversion
ggplot(data.aggr, aes_string(x=Faktor, y="Freq", group=1)) + geom_line(size=0.8) + geom_point(size=3) + geom_point(size=1, colour="white") + theme_bw() + ylim(0, NA) + ylab("Häufigkeit")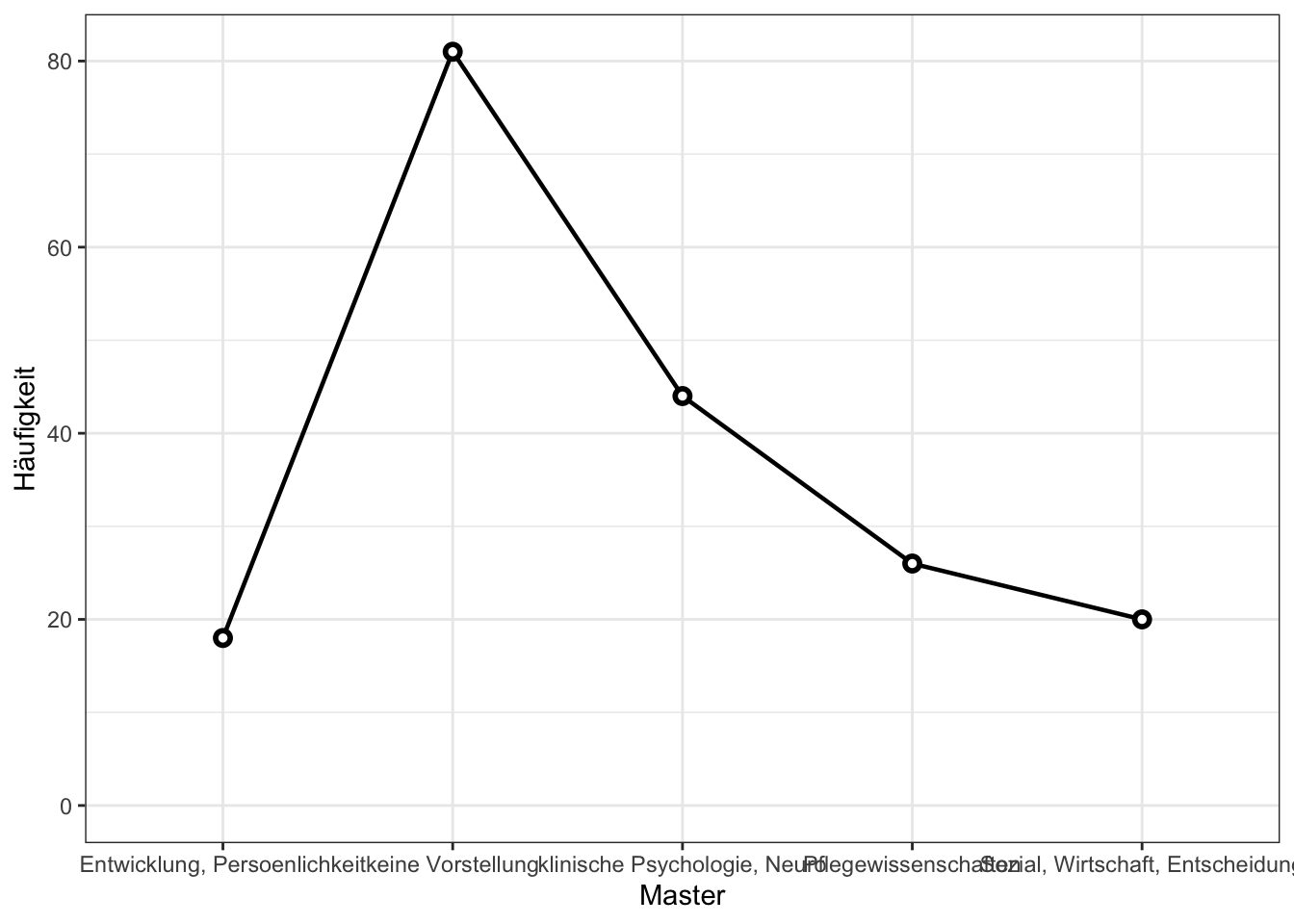
# Farbige Version
ggplot(data.aggr, aes_string(x=Faktor, y="Freq", group=1)) + geom_line(size=0.8, colour="red") + geom_point(size=3, colour="red") + geom_point(size=1, colour="white") + theme_grey() + ylim(0, NA) + ylab("Häufigkeit")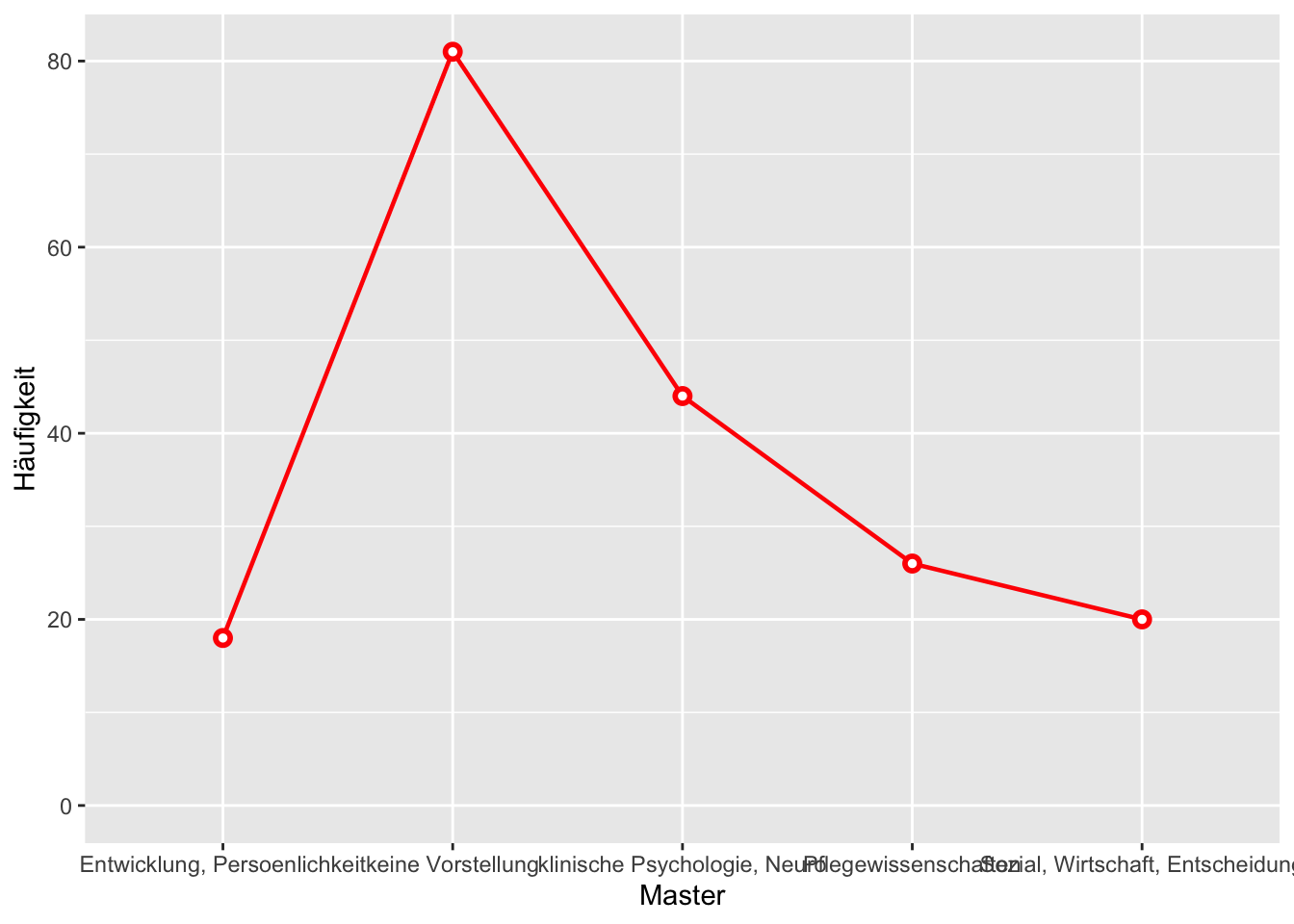
Hinweis: Vertikale Beschriftung der Abszisse
last_plot() + theme(axis.text.x = element_text(angle=90, vjust=0.5, hjust=1))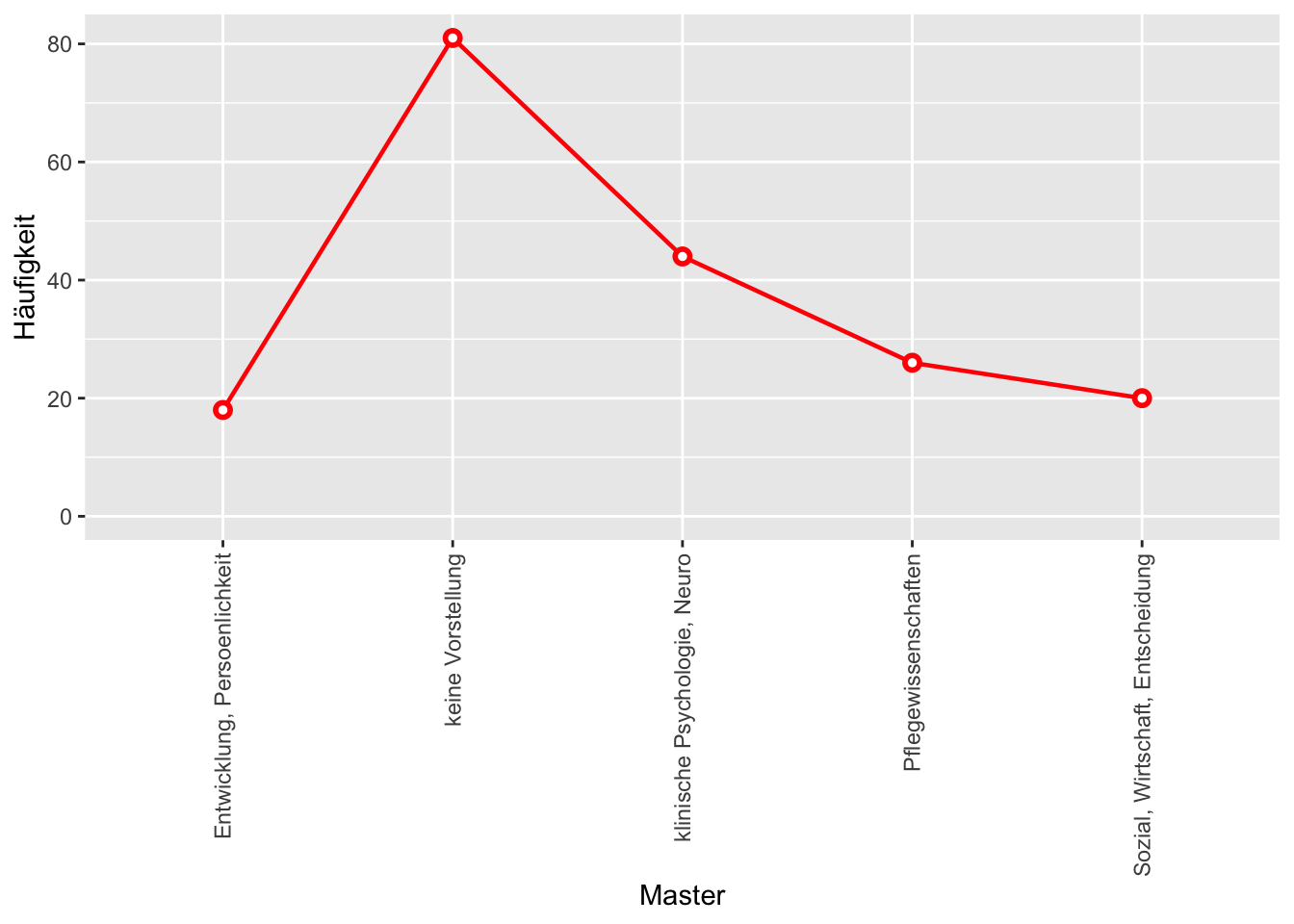
Zweifaktoriell
Datensatz einlesen und Variablen spezifizieren
# Datensatz 'data': pro Versuchsperson eine Zeile
# Häufigkeitstabellen zuerst umwandeln, vgl. Umwandlung von Datenstrukturen
Master <- rep(c("Entwicklung, Persoenlichkeit", "keine Vorstellung", "klinische Psychologie, Neuro", "Pflegewissenschaften", "Sozial, Wirtschaft, Entscheidung", "Entwicklung, Persoenlichkeit", "keine Vorstellung", "klinische Psychologie, Neuro", "Pflegewissenschaften", "Sozial, Wirtschaft, Entscheidung"), c(3,15,4,5,5,15,66,40,21,14))
Geschlecht <- rep(c("männlich", "weiblich"), c(32,156))
data <- data.frame(Master, Geschlecht)
# Variablen spezifizieren
Faktor1 <- "Master" # Bezeichnung des Faktors auf der Abszisse eingeben
Faktor2 <- "Geschlecht" # Bezeichnung des Faktors für die Linienfarbe eingebenLiniendiagramm
data2 <- na.omit(data[, c(Faktor1, Faktor2), drop=FALSE]); library(ggplot2)
data.aggr <- data.frame(xtabs(paste("~", Faktor1, "+", Faktor2), data2))
# Schwarzweissversion
ggplot(data.aggr, aes_string(x=Faktor1, y="Freq", group=Faktor2, shape=Faktor2)) + geom_line(size=0.8) + geom_point(size=3) + theme_bw()+ ylab("Häufigkeit") + ylim(0, NA)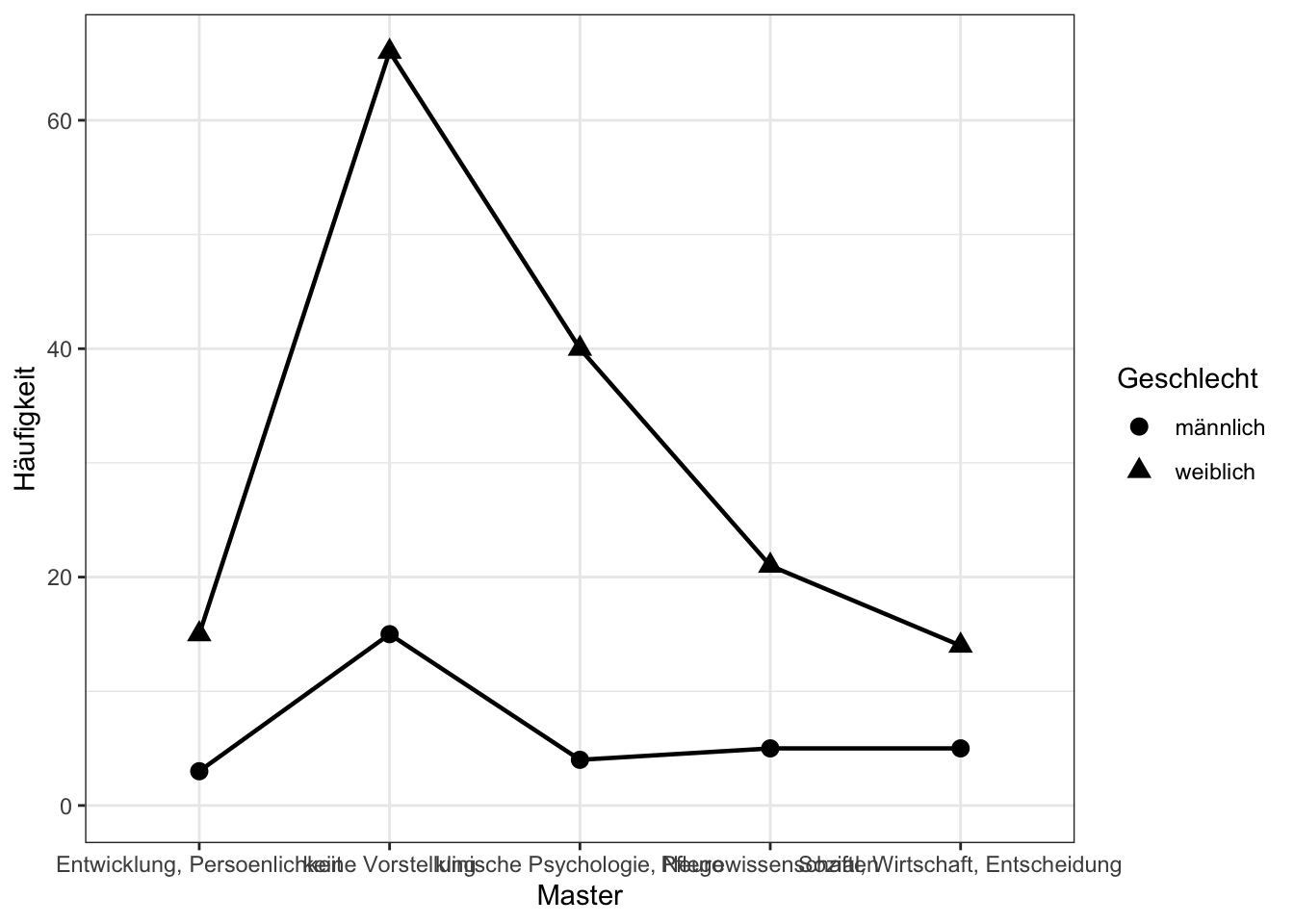
# Farbige Version
ggplot(data.aggr, aes_string(x=Faktor1, y="Freq", group=Faktor2, shape=Faktor2, colour=Faktor2)) + geom_line(size=0.8) + geom_point(size=3) + theme_grey() + ylab("Häufigkeit") + ylim(0, NA)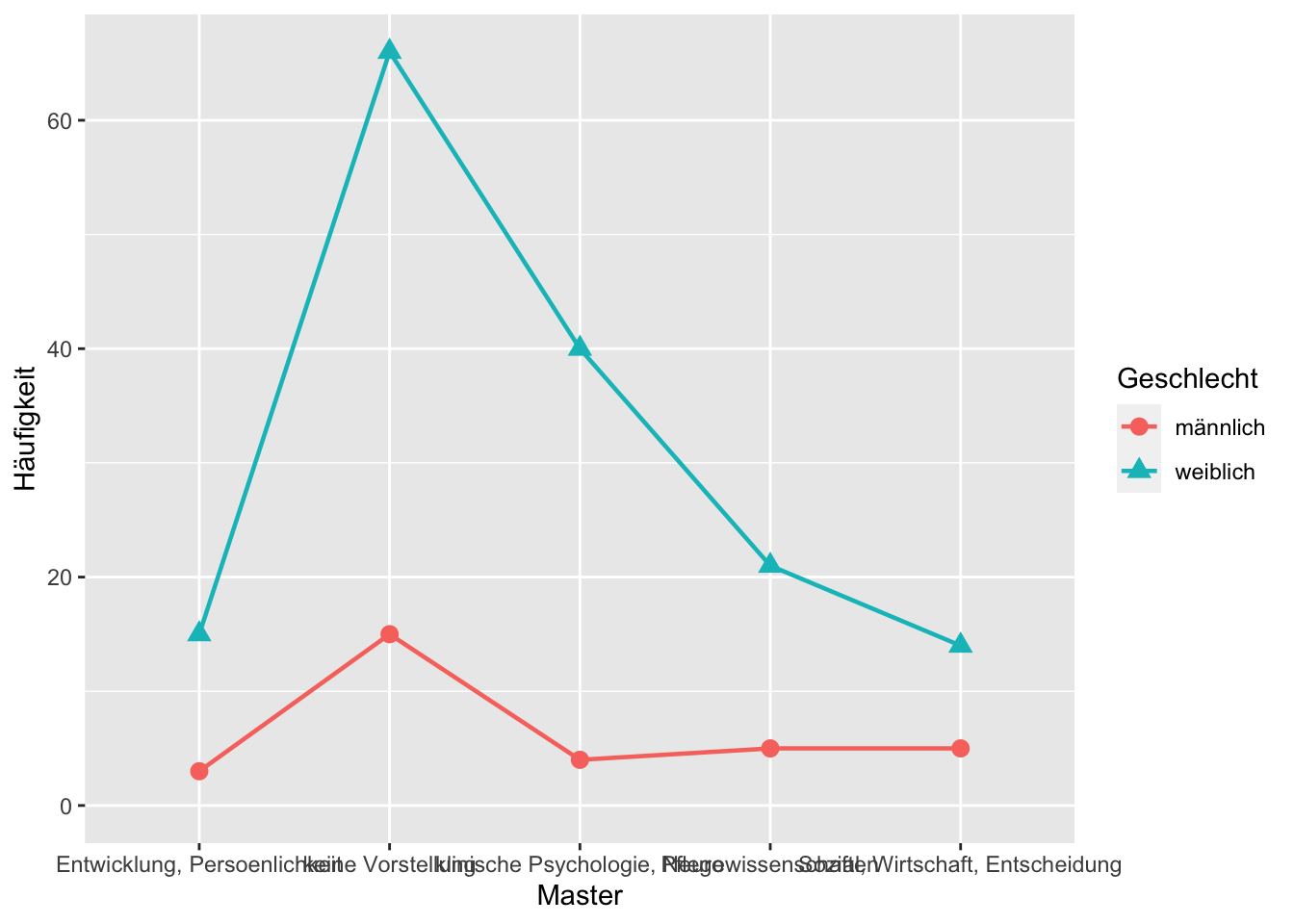
Hinweis: Vertikale Beschriftung der Abszisse
last_plot() + theme(axis.text.x = element_text(angle=90, vjust=0.5, hjust=1))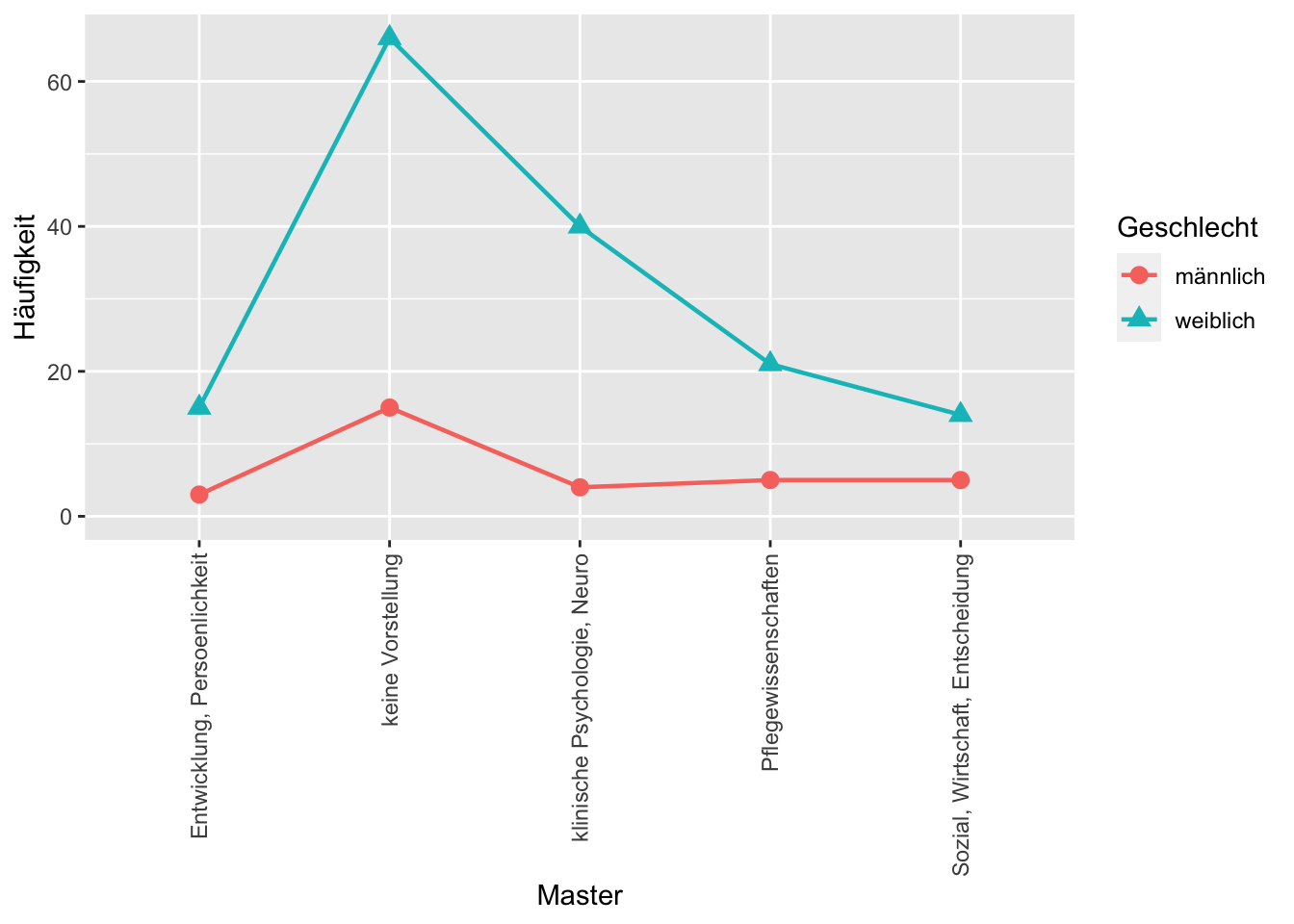
Dreifaktoriell
Datensatz einlesen und Variablen spezifizieren
# Datensatz 'data': pro Versuchsperson eine Zeile
# Häufigkeitstabellen zuerst umwandeln, vgl. Umwandlung von Datenstrukturen
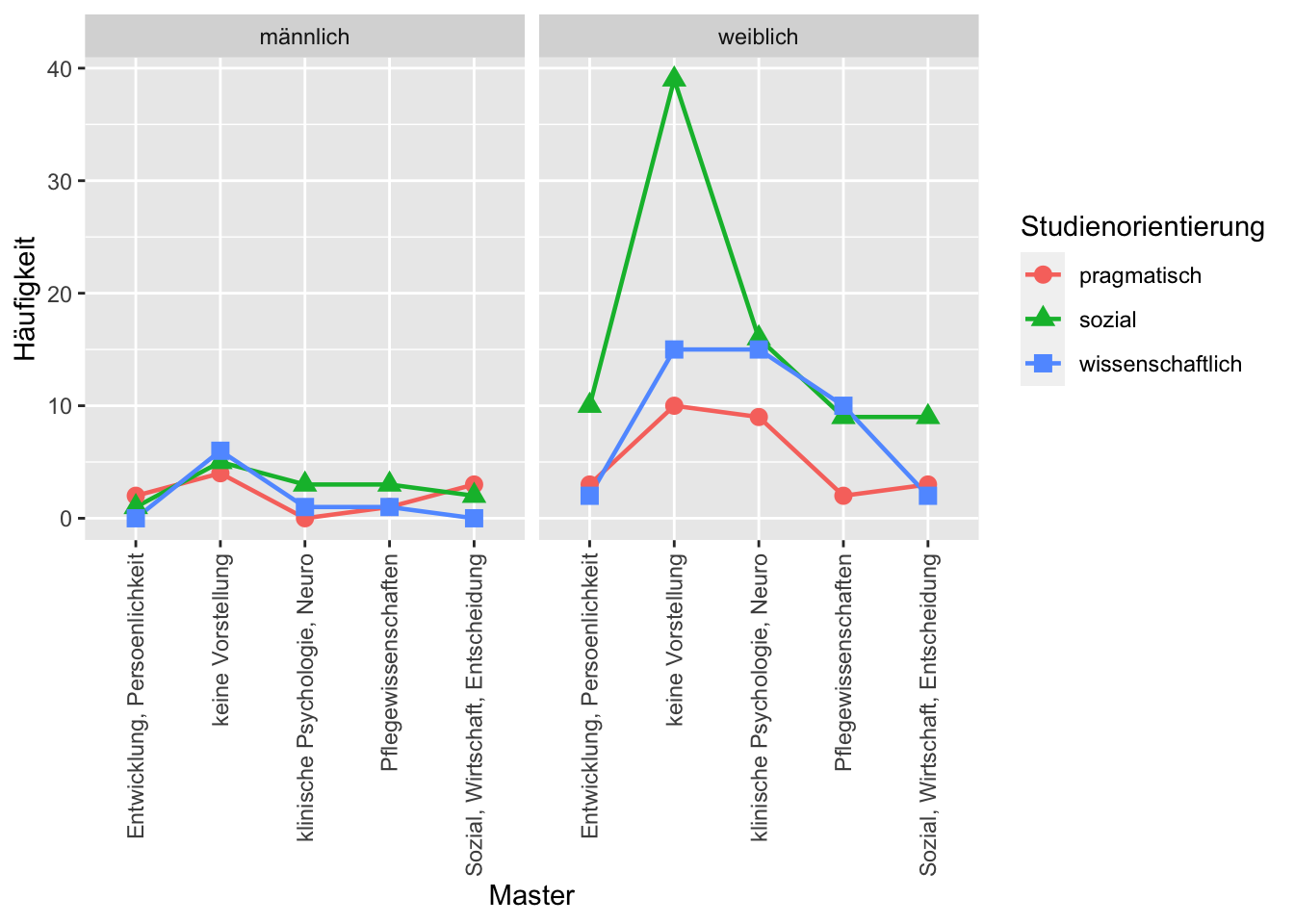
Master <- rep(rep(c("Entwicklung, Persoenlichkeit", "keine Vorstellung", "klinische Psychologie, Neuro", "Pflegewissenschaften", "Sozial, Wirtschaft, Entscheidung"), 6), c(2,4,0,1,3,1,5,3,3,2,0,6,1,1,0,3,10,9,2,3,10,39,16,9,9,2,15,15,10,2))
Studienorientierung <- rep(rep(c("pragmatisch", "sozial", "wissenschaftlich"), each=5, len=30), c(2,4,0,1,3,1,5,3,3,2,0,6,1,1,0,3,10,9,2,3,10,39,16,9,9,2,15,15,10,2))
Geschlecht <- rep(rep(c("männlich", "weiblich"), c(15,15)), c(2,4,0,1,3,1,5,3,3,2,0,6,1,1,0,3,10,9,2,3,10,39,16,9,9,2,15,15,10,2))
data <- data.frame(Master, Studienorientierung, Geschlecht)
# Variablen spezifizieren
Faktor1 <- "Master" # Name des ersten Faktors eingeben (Abszisse)
Faktor2 <- "Studienorientierung" # Name des zweiten Faktors eingeben (Linienfarbe)
Faktor3 <- "Geschlecht" # Unterteilung der PlotsLiniendiagramm
data2 <- na.omit(data[, c(Faktor1, Faktor2, Faktor3), drop=FALSE]); library(ggplot2)
data.aggr <- data.frame(xtabs(paste("~", Faktor1, "+", Faktor2, "+", Faktor3), data2))
# Schwarzweissversion
ggplot(data.aggr, aes_string(x=Faktor1, y="Freq", group=Faktor2, shape=Faktor2)) + geom_line(size=0.8) + geom_point(size=3) + facet_grid(paste("~", Faktor3)) + theme_bw() + ylab("Häufigkeit") + ylim(0, NA)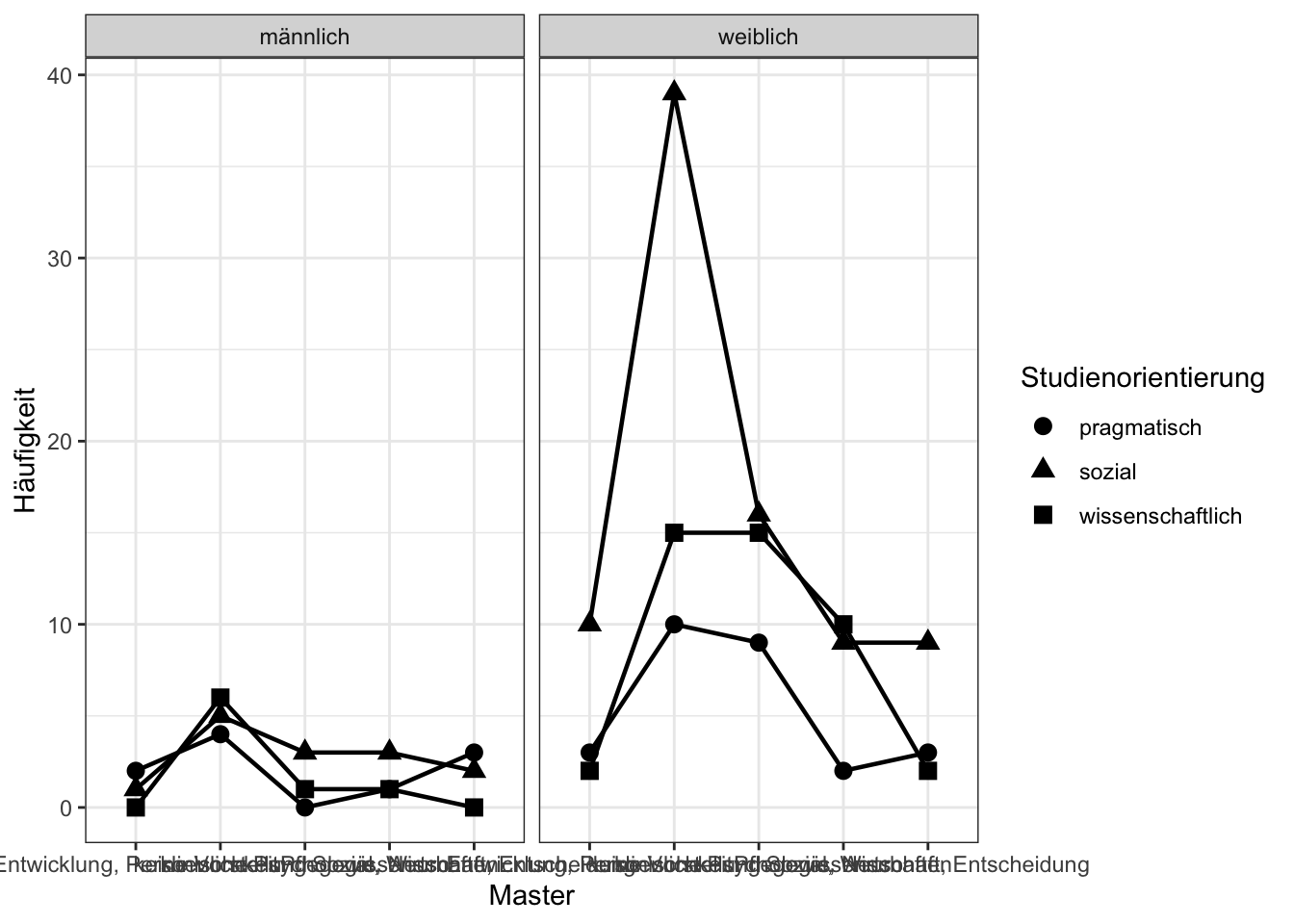
# Farbige Version
ggplot(data.aggr, aes_string(x=Faktor1, y="Freq", group=Faktor2, shape=Faktor2, colour=Faktor2)) + geom_line(size=0.8) + geom_point(size=3) + facet_grid(paste("~", Faktor3)) + theme_grey() + ylab("Häufigkeit") + ylim(0, NA)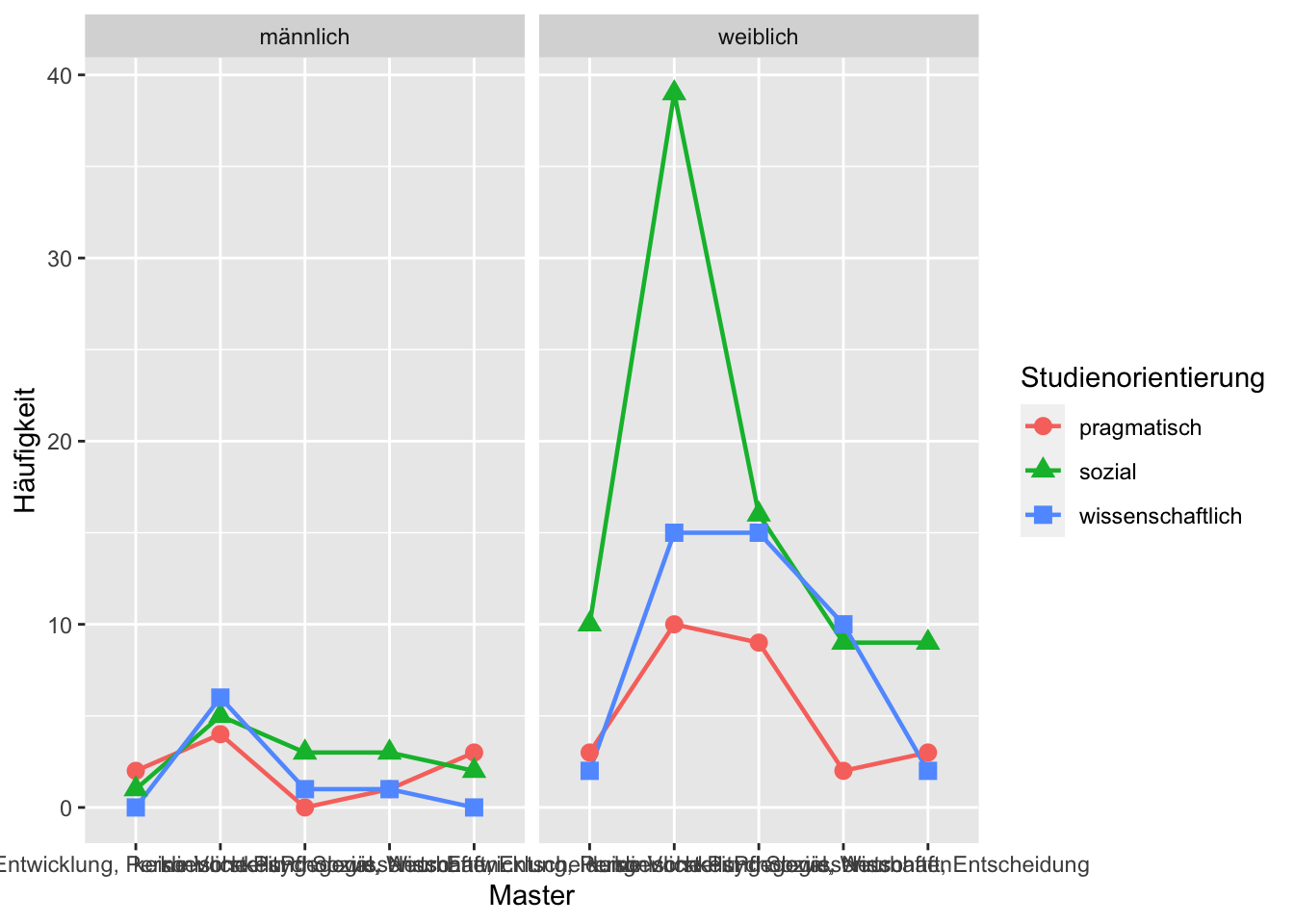
Hinweis: Vertikale Beschriftung der Abszisse
last_plot() + theme(axis.text.x = element_text(angle=90, vjust=0.5, hjust=1))