Diskriminanzanalyse
- Erforderliche Pakete laden
- Datensatz einlesen und Modell spezifizieren
- Diskriminanzanalyse
- Gruppenspezifische Verteilungen und Streudiagramme
- Univariate und multivariate Ausreisser
- Überprüfen der Gleichheit der Kovarianzmatrizen
- Modellstabilität (leave-one-out cross-validation)
- Wichtigkeit der Prädiktoren für die Klassifikation
- Darstellung der Fälle im Diskriminanzfunktionsraum
- ROC-Analyse
Erforderliche Pakete laden
library(MASS) # Diskriminanzanalyse
library(candisc) # Diskriminanzanalyse und Grafik
library(klaR) # Wichtigkeit der Prädiktoren für die KLassifikation, Klassifikationsplots
library(ggplot2) # Grafik
library(GGally) # Grafik
library(pROC) # ROC-AnalyseDatensatz einlesen und Modell spezifizieren
# Dateneingabe
data<-read.table(file="https://mmi.psycho.unibas.ch/r-toolbox/data/Diskriminanzanalyse/lda4.txt", header=TRUE)
data$workstat[data$workstat=="0"] <- NA; data$control[data$control==0] <- NA; data$attmar[data$attmar==0] <- NA; data$attrol[data$attrol==0] <- NA; data$atthouse[data$atthouse==1] <- NA
data$log_control <- log10(data$control)
data$log_attmar <- log10(data$attmar)
# Modell spezifizieren
model <- workstat~log_control+log_attmar+attrole+atthouse
# Für die Klassifikation zu verwendende a priori Wahrscheinlichkeiten:
# Wenn Sie aus den Daten berechnet werden sollen: "from.data"
# Wenn sie für alle Gruppen gleich sein sollen: "equal"
# Für andere Varianten ist der Vektor mit den gruppenspezifischen Wahrscheinlichkeiten einzugeben.
apriori <- "from.data"Diskriminanzanalyse
Um einen möglichst vollständigen Output zu erhalten, wurden die Ergebnisse der Funktionen lda() und candisc() kombiniert.
Hinweise zum Output
Für das Modell mit einer zweistufigen Gruppenvariable und zwei Prädiktoren liefert die Funktion lda() den Signifikanztest für die Diskriminanzfunktion nicht. Sie erhalten ihn mit: anova(he.mod, test=“Wilks”)
Wenn das Modell nur einen Prädiktor enthält, liefert die Funktion candisc() eine Fehlermeldung. Aus diesem Grund beschränken wir uns in diesem Fall auf den Output der Funktion lda(). Der Signifikanztest für das Modell entpricht der ANOVA für den Prädiktor.
library(MASS); library(candisc)
# Modellschätzung
variablen <- all.vars(model)
data[,variablen[1]] <- as.factor(data[,variablen[1]]) # Die Zielvariable als Faktor definieren
data2 <- na.omit(data[, variablen, drop=FALSE])
# Varianzanalysen
ANOVA <- list()
for (i in variablen[-1]) {
model2 <- paste0(i, "~", variablen[1])
ANOVA[[i]] <- data.frame(anova(lm(model2, data2)))}
if (class(apriori)=="numeric") {
prior <- apriori
} else if (apriori=="equal") {
prior <- rep(1,nlevels(data2[,variablen[1]]))/nlevels(data2[,variablen[1]])
} else prior <- table(data2[,variablen[1]])/nrow(data2)
m <- lda(model, data2, prior=as.numeric(prior))
if (length(variablen[-1])>1) {
# Kanonische Korrelationskoeffienten im Quadrat, Eigenwerte,
# Anteile der durch die Diskriminanzfunktion erklärten Varianz, Inferenzstatistik
model2 <- paste("cbind(",paste(variablen[-1], collapse=","),")~",variablen[1],sep="")
he.mod <- lm(model2, data2)
he.can <- candisc(he.mod)}
# Klassifikationstabelle
pr <- predict(m); klass.tab <- xtabs(~ data2[,variablen[1]] + pr$class)
# Nach Zufall zu erwartender Anteil richtiger Klassifikationen
Zufall <- as.numeric(rowSums(klass.tab) %*% rowSums(klass.tab)/sum( (klass.tab))^2)
# Anteil richtig klassifiziert
ant.ri.kl <- mean(data2[,variablen[1]]==pr$class) # insgesamt
Prozent.korrekt <- diag(klass.tab)/apply(klass.tab,1,sum)*100 # pro Zeile
klass.tab <-cbind(klass.tab, "korrekt in %"=Prozent.korrekt)
names(dimnames(klass.tab)) <- c("beobachtet", "vorhergesagt")
# Output
if (length(variablen[-1])>1) {
list("Gruppenmittelwerte der Prädiktoren"=m$means, ANOVAs=ANOVA, "Kanonische Korrelationskoeffienten im Quadrat, Eigenwerte, Anteile der durch die Diskriminanzfunktion erklärten Varianz, Inferenzstatistik"=he.can, "Unstandardisierte kanonische Parameter" = he.can$coeffs.raw,
"Standardisierte kanonische Parameter" = he.can$coeffs.std, "Kanonische Strukturkoeffizienten (Ladungen)" = he.can$structure, "Koordinaten der Zentroide" = aggregate(as.formula(paste(".~", variablen[1])), he.can$scores, mean), Klassifikation=klass.tab, "A priori Wahrscheinlichkeiten"=m$prior, "Anteil richtig klassifizierter Fälle"=ant.ri.kl,
"Nach Zufall zu erwartender Anteil richtig klassifizierter Fälle"=Zufall)
} else {
list("Gruppenmittelwerte des Prädiktors"=m$means, ANOVAs=ANOVA, "Unstandardisierte kanonische Parameter" = m$scaling, Klassifikation=klass.tab, "A priori Wahrscheinlichkeiten"=m$prior, "Anteil richtig klassifizierter Fälle"=ant.ri.kl,
"Nach Zufall zu erwartender Anteil richtig klassifizierter Fälle"=Zufall)
} ## $`Gruppenmittelwerte der Prädiktoren`
## log_control log_attmar attrole atthouse
## role.dissatisfied.housewives 0.8414321 1.375472 35.66667 24.92593
## role.satisfied.housewives 0.8135600 1.293481 37.19118 22.50735
## working 0.8191829 1.341147 33.92531 23.63071
##
## $ANOVAs
## $ANOVAs$log_control
## Df Sum.Sq Mean.Sq F.value Pr..F.
## workstat 2 0.04204707 0.021023536 3.270224 0.03889295
## Residuals 455 2.92509328 0.006428776 NA NA
##
## $ANOVAs$log_attmar
## Df Sum.Sq Mean.Sq F.value Pr..F.
## workstat 2 0.3744977 0.18724886 8.190523 0.000320211
## Residuals 455 10.4020499 0.02286165 NA NA
##
## $ANOVAs$attrole
## Df Sum.Sq Mean.Sq F.value Pr..F.
## workstat 2 948.4307 474.21535 10.83299 2.534393e-05
## Residuals 455 19917.6850 43.77513 NA NA
##
## $ANOVAs$atthouse
## Df Sum.Sq Mean.Sq F.value Pr..F.
## workstat 2 302.5046 151.25230 7.682769 0.0005230189
## Residuals 455 8957.6810 19.68721 NA NA
##
##
## $`Kanonische Korrelationskoeffienten im Quadrat, Eigenwerte, Anteile der durch die Diskriminanzfunktion erklärten Varianz, Inferenzstatistik`
##
## Canonical Discriminant Analysis for workstat:
##
## CanRsq Eigenvalue Difference Percent Cumulative
## 1 0.062974 0.067206 0.030669 64.781 64.781
## 2 0.035249 0.036537 0.030669 35.219 100.000
##
## Test of H0: The canonical correlations in the
## current row and all that follow are zero
##
## LR test stat approx F numDF denDF Pr(> F)
## 1 0.90400 5.8489 8 904 2.628e-07 ***
## 2 0.96475 5.5171 3 453 0.0009964 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## $`Unstandardisierte kanonische Parameter`
## Can1 Can2
## log_control 2.00403954 -4.3327757
## log_attmar 3.59667506 -0.9335778
## attrole -0.08676832 -0.1245645
## atthouse 0.06199950 -0.1338532
##
## $`Standardisierte kanonische Parameter`
## Can1 Can2
## log_control 0.1606832 -0.3474004
## log_attmar 0.5438197 -0.1411576
## attrole -0.5740833 -0.8241531
## atthouse 0.2750935 -0.5939101
##
## $`Kanonische Strukturkoeffizienten (Ladungen)`
## Can1 Can2
## log_control 0.3062913 -0.4841685
## log_attmar 0.7063987 -0.3072306
## attrole -0.7044050 -0.6348437
## atthouse 0.6384616 -0.4455330
##
## $`Koordinaten der Zentroide`
## workstat Can1 Can2
## 1 role.dissatisfied.housewives 0.2391324 -0.3712843
## 2 role.satisfied.housewives -0.3938458 -0.0401426
## 3 working 0.1418809 0.1474416
##
## $Klassifikation
## vorhergesagt
## beobachtet role.dissatisfied.housewives
## role.dissatisfied.housewives 3
## role.satisfied.housewives 2
## working 2
## vorhergesagt
## beobachtet role.satisfied.housewives working korrekt in %
## role.dissatisfied.housewives 11 67 3.703704
## role.satisfied.housewives 33 101 24.264706
## working 31 208 86.307054
##
## $`A priori Wahrscheinlichkeiten`
## role.dissatisfied.housewives role.satisfied.housewives
## 0.1768559 0.2969432
## working
## 0.5262009
##
## $`Anteil richtig klassifizierter Fälle`
## [1] 0.5327511
##
## $`Nach Zufall zu erwartender Anteil richtig klassifizierter Fälle`
## [1] 0.3963406Gruppenspezifische Verteilungen und Streudiagramme
library(ggplot2); library(GGally)
ggally_mysmooth <- function(data, mapping, ...){
ggplot(data = data, mapping=mapping) +
geom_density(mapping = aes_string(colour=variablen[1]), fill=NA)}
ggpairs(data2, columns=match(variablen[-1],names(data2)), diag = list(continuous = ggally_mysmooth), ggplot2::aes_string(colour=variablen[1]))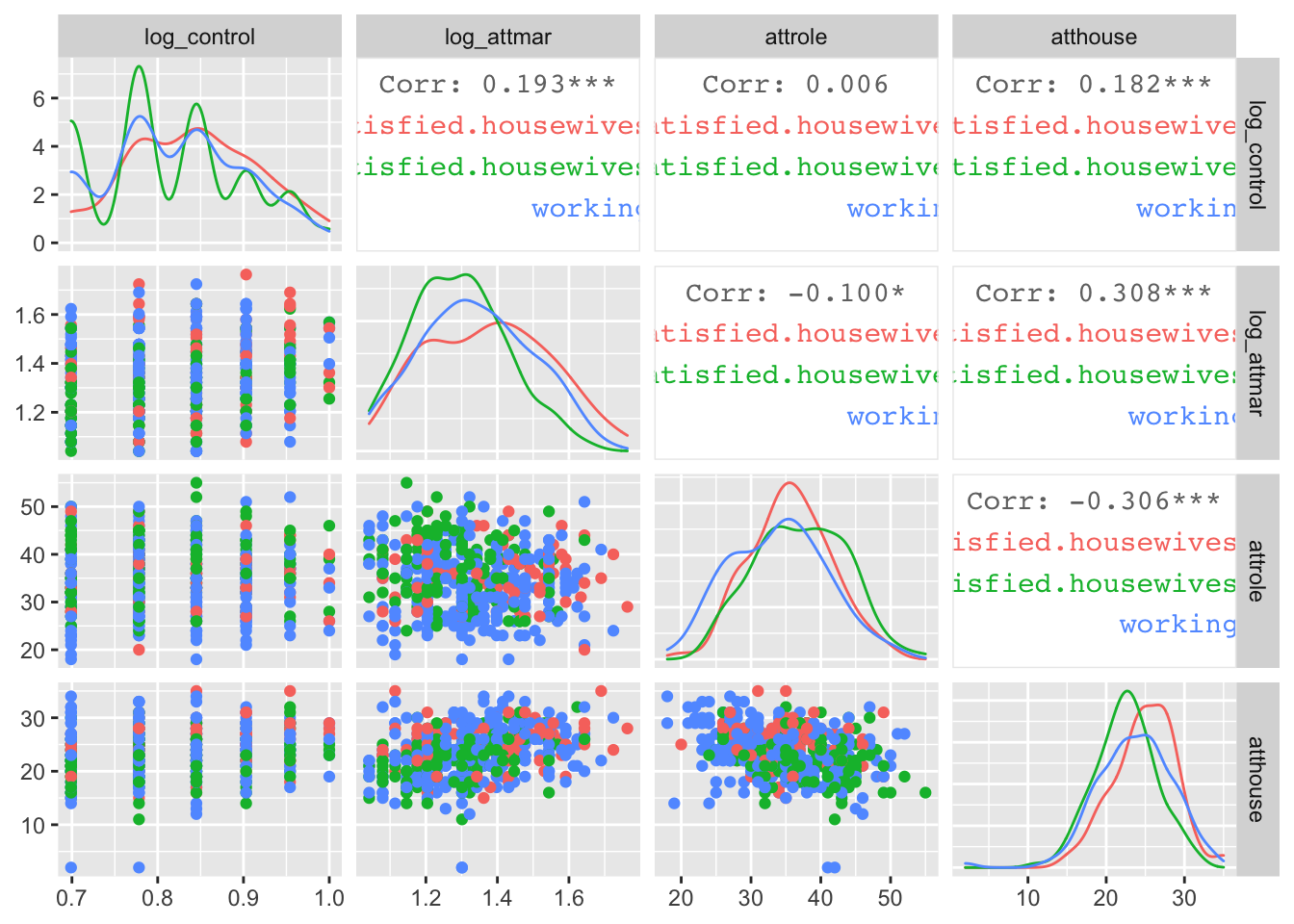
Univariate und multivariate Ausreisser
Univariate Ausreisser pro Prädiktor und Gruppe anhand des Boxplotkriteriums
Es werden die z-Werte mit den darüberstehenden Zeilennamen der Ausreisser ausgegeben. Die Plots können ignoriert werden.
UV <- variablen[-1] # Vektor mit den Namen der Prädiktoren
AV <- variablen[1] # Name der Zielvariable
data2[,AV] <- as.factor(data2[,AV]) # Zielvariable als Faktor definieren
library(car)
z.werte <- numeric()
for (i in levels(data2[,AV])){
data.pred <- data2[data2[,AV]==i, UV, drop=FALSE] # Datensatz mit den zu transformierenden Variablen
data.z <- scale(data.pred); rownames(data.z) <- rownames(data.pred) # z-Transformation
for (k in UV) {
model2 <- as.formula(paste0("~", k))
ar <- sort(as.numeric(Boxplot(model2, data=data.z, range=1.5, id.n=Inf)))
z <- data.z[,k][rownames(data.z) %in% ar]
z.werte <- c(z.werte, z)
}}
z.werte## 35 27 52 54 73 131 140 154
## -2.507519 1.682082 1.682082 2.229185 1.682082 1.682082 1.682082 2.229185
## 177 203 423 435 261 299
## 1.682082 1.682082 2.229185 -2.963154 -4.450987 -4.450987Multivariate Ausreisser pro Gruppe
Es werden die quadrierten Mahalanobisdistanzen mit den darüberstehenden Zeilennamen der Ausreisser ausgegeben
Nicht sinnvoll, wenn das Modell nur einen Prädiktor enthält (Fehlermeldung).
UV <- variablen[-1] # Vektor mit den Namen der Prädiktoren
AV <- variablen[1] # Name der Zielvariable
kw <- qchisq(p=.001, df=length(UV), lower.tail=FALSE) # kritischer Wert
data2[,AV] <- as.factor(data2[,AV]) # Zielvariable als Faktor definieren
data3 <- split(data2, data2[,AV]) # Datensatz nach den Stufen der Zielvariablen aufteilen
library(heplots)
mahal <- numeric(); namen <- character()
# Zeilennamen und quadrierte Mahalanobisdistanzen
for (i in levels(data2[,AV])){
data.pred <- data2[data2[,AV]==i, UV, drop=FALSE]
md <- Mahalanobis(data.pred)
mahal <- c(mahal, md)
namen <- c(namen, rownames(data.pred))
}
names(mahal) <- namen
mahal[mahal>kw]## 261 299
## 22.27617 20.88468Ausschliessen von Ausreissern anhand ihrer Zeilennamen
In unserem Beispiel sind die Probandinnen mit den Zeilennamen 261 und 299 Ausreisser: data2 <- data2[!(rownames(data2) %in% c(“261”, “299”)),]
Anschliessend kann man die Diskriminanzanalyse ohne diese Ausreisser rechnen, indem man den Code von “# Varianzanalysen” an kopiert und ausführt.
Überprüfen der Gleichheit der Kovarianzmatrizen
Box’s M Test
Nicht sinnvoll, wenn das Modell nur einen Prädiktor enthält. Es erscheint eine Fehlermeldung.
library(heplots)
boxM(data2[,variablen[-1]], data2[,variablen[1]])##
## Box's M-test for Homogeneity of Covariance Matrices
##
## data: data2[, variablen[-1]]
## Chi-Sq (approx.) = 42.44, df = 20, p-value = 0.002423Modellstabilität (leave-one-out cross-validation)
# Klassifikationstabelle
pr2 <- lda(model, data2, CV=TRUE)
klass.tab2 <- xtabs(~ data2[,variablen[1]] + pr2$class)
# Anteil richtig klassifiziert
ant.ri.kl2 <- sum(diag(klass.tab2))/sum(klass.tab2)
Prozent.korrekt2 <- diag(klass.tab2)/apply(klass.tab2,1,sum)*100
klass.tab2 <-cbind(klass.tab2, "korrekt in %"=Prozent.korrekt2)
names(dimnames(klass.tab2)) <- c("beobachtet", "vorhergesagt")
list(Klassifikation = klass.tab2, "Anteil richtig klassifizierter Fälle" = ant.ri.kl2)## $Klassifikation
## vorhergesagt
## beobachtet role.dissatisfied.housewives
## role.dissatisfied.housewives 2
## role.satisfied.housewives 2
## working 2
## vorhergesagt
## beobachtet role.satisfied.housewives working korrekt in %
## role.dissatisfied.housewives 11 68 2.469136
## role.satisfied.housewives 30 104 22.058824
## working 32 207 85.892116
##
## $`Anteil richtig klassifizierter Fälle`
## [1] 0.5218341Wichtigkeit der Prädiktoren für die Klassifikation
library(klaR)
res <- stepclass(model, data = data2, method="lda", direction="both")## `stepwise classification', using 10-fold cross-validated correctness rate of method lda'.## 458 observations of 4 variables in 3 classes; direction: both## stop criterion: improvement less than 5%.## correctness rate: 0.53493; in: "attrole"; variables (1): attrole
##
## hr.elapsed min.elapsed sec.elapsed
## 0.00 0.00 0.16plot(res)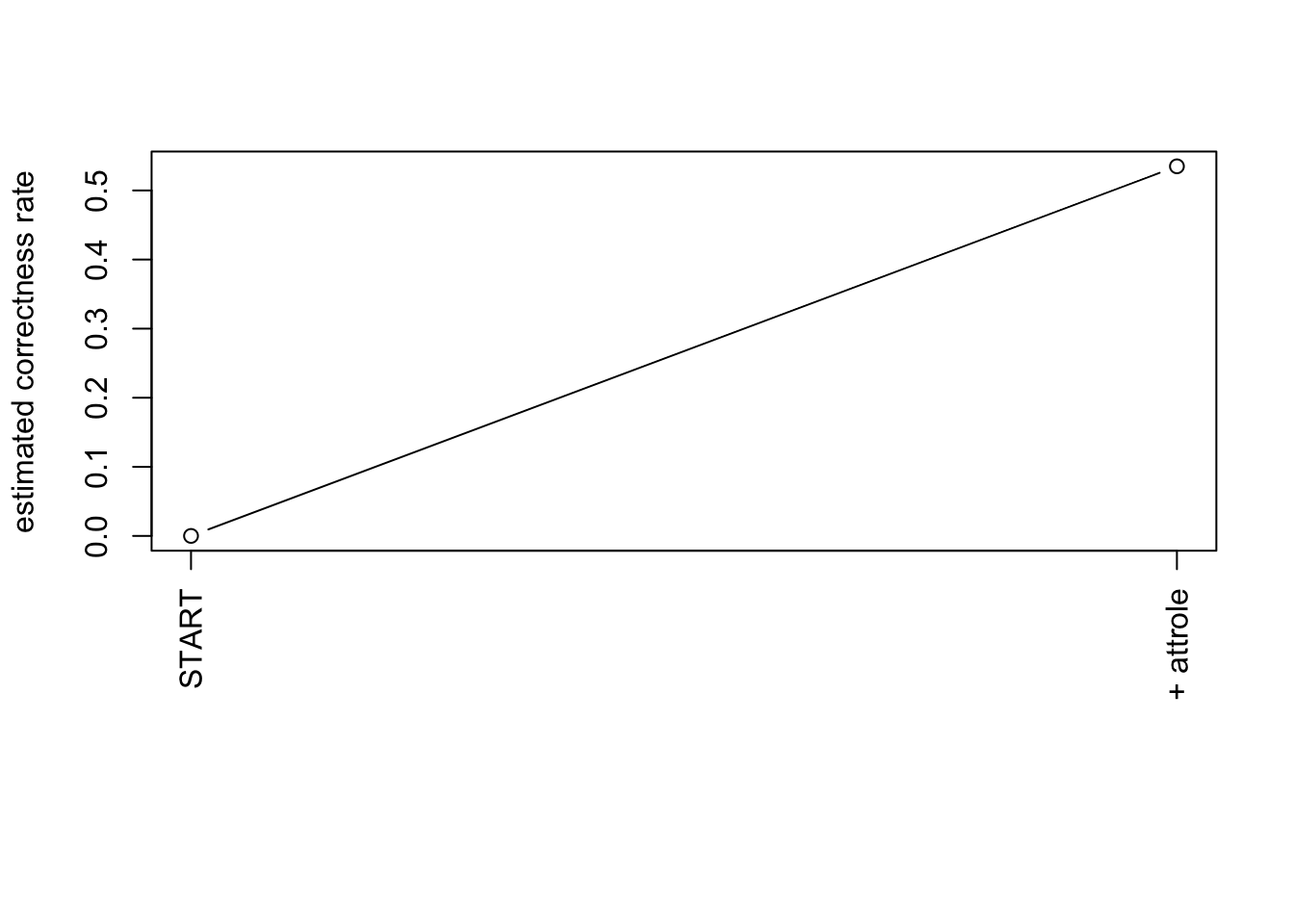
Darstellung der Fälle im Diskriminanzfunktionsraum
Mehr als ein Prädiktor
Wenn nur eine Diskriminanzfunktion vorhanden ist, werden die gruppenspezifischen Boxplots der Diskriminanzfunktionswerte sowie ein Strukturplot mit den Ladungen der Prädiktoren dargestellt.
Wenn mindestens zwei Diskriminanzfunktionen vorhanden sind, wird ein Streudiagramm mit den Zentroiden und den Ladungsvektoren für die ersten beiden Diskriminanzfunktionen dargestellt.
plot(he.can)## Vector scale factor set to 5.101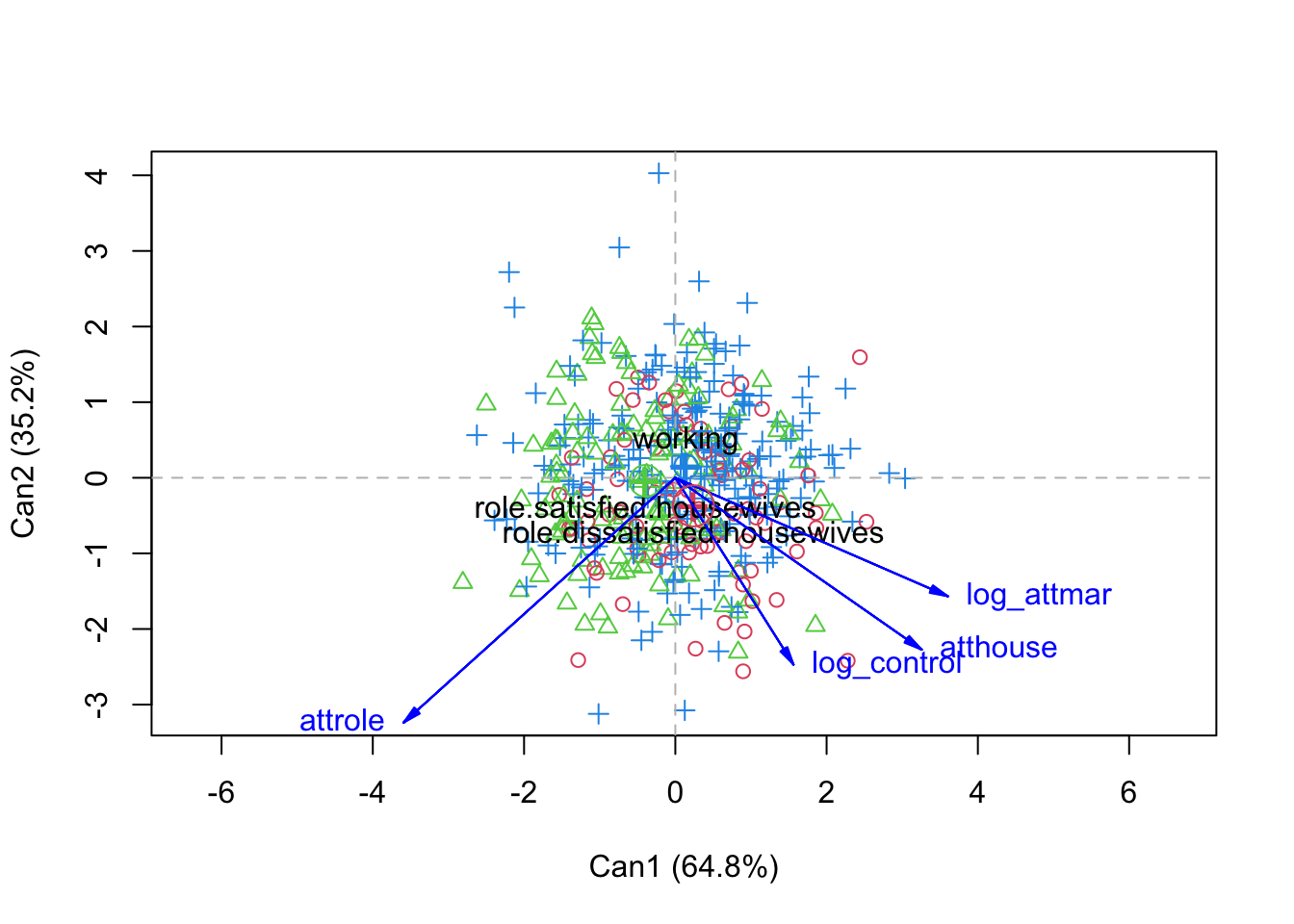
Nur ein Prädiktor
Gruppenspezifische Histogramme der Diskriminanzfunktion. Für mehr als zwei Prädiktoren liefert plot(m) eine schlechtere Variante des obigen Plots.
# plot(m)ROC-Analyse
Wenn die Zielvariable zweistufig ist, kann eine ROC-Analyse gerechnet werden.
Üblicherweise werden die “cases” (positive Diagnose) mit 1 und die “controls” (negative Diagnose) mit 0 kodiert. Wenn man andere Kodierungen der Stufen verwendet, werden die Probanden der zweiten Stufe der Zielvariable als “cases” betrachtet. Die Fläche unter der Kurve wird durch die Wahl der Kategorie für die “cases” nicht beeinflusst, der Verlauf der ROC-Kurve ändert jedoch, da die Achsen vertauscht werden.
Mehr als ein Prädiktor
# library(pROC)
# data2$Can1 <- he.can$scores[,-1]
# ROC <- roc(response=data2[,variablen[1]], predictor=data2$Can1, ci=TRUE); ROC
# plot(ROC, main="ROC-Kurve"); grid()
# plot.roc(smooth(ROC), add=TRUE, lty=2)Nur ein Prädiktor
# library(pROC)
# ROC <- roc(response=data2[,variablen[1]], predictor=data2[,variablen[-1]], ci=TRUE); ROC
# plot(ROC, main="ROC-Kurve"); grid()
# plot.roc(smooth(ROC), add=TRUE, lty=2)