Einfache und multple Korrespondenzanalyse
Skript mit Erklärung der Verfahren und Vorgehensweisen anhand von Beispielen.
Erforderliche Pakete laden
if (!require(ca)) {install.packages("ca")} # Datensatz
if (!require(FactoMineR)) {install.packages("FactoMineR")} # Korrespondenzanalyse
if (!require(factoextra)) {install.packages("factoextra")} # GrafikenEinfache Korrespondenzanalyse
Datensatz einlesen und Variablen spezifizieren
Die Datensätze können drei verschiedene Strukturen aufweisen.
1) Man kann die Daten in EXCEL als Kreuztabelle eingeben. In der ersten Spalte lässt man die erste Zelle leer. Darunter stehen die Stufenbezeichnungen des Zeilenfaktors. In den folgenden Spalten stehen zuoberst die Stufenbezeichnungen des Spaltenfaktors und darunter die Zellhäufigkeiten.
2) Pro Proband eine Zeile
3) Zwei Spalten mit den Faktorstufenkombinationen und eine Spalte mit den Häufigkeiten pro Faktorstufenkombination
# Datensatz
data <- margin.table(HairEyeColor, 1:2)
# Variablen spezifizieren
# Datenformat: 1=Kontingenztabelle, 2=eine Zeile pro Proband,
# 3=Zwei Spalten mit den Faktorstufenkombinationen und eine Spalte mit den Häufigkeiten pro Faktorstufenkombination
Datenformat <- 1
Faktor1 <- "Hair" # Name des Zeilenfaktors der Kontingenztabelle
Faktor2 <- "Eye" # Name des Spaltenfaktors der Kontingenztabelle
Häufigkeiten <- "Häufigkeit" # Spalte mit den Häufigkeiten für Datenformat 3
suprow <- NULL # Nummern der zusätzlichen passiven Zeilen (passiv). Falls keine: suprow <- NULL
supcol <- NULL # Nummern der zusätzlichen passiven Spalten (passiv). Falls keine: supcol <- NULLHäufigkeitstabelle und Korrespondenzanalyse
# Kontingenztabelle
if (Datenformat ==1) {
tab <- as.matrix(data)
#Name des Zeilen- und Spaltenfaktors einfügen
dimnames(tab) <- eval(parse(text=paste0("list(", Faktor1, " = rownames(tab), ", Faktor2, " = colnames(tab))")))}
if (Datenformat ==2) tab <- xtabs(paste("~", Faktor1, "+", Faktor2), data)
if (Datenformat ==3) tab <- xtabs(as.formula(paste(Häufigkeiten, "~", Faktor1, "+", Faktor2)), data=data)
# Zeilenweise relative Häufigkeiten
tab.rel.zeilen <- prop.table(tab, 1)
# Spaltenweise relative Häufigkeiten
tab.rel.spalten <- prop.table(tab, 2)
# Korrespondenzanalyse
library(FactoMineR)
res <- CA(tab, row.sup=suprow, col.sup=supcol, graph=FALSE)
# Ergebnisse
list(Häufigkeitstabelle=tab, "Zeilenweise relative Häufigkeiten"=tab.rel.zeilen, "Spaltenweise relative Häufigkeiten"=tab.rel.spalten); summary(res)## $Häufigkeitstabelle
## Eye
## Hair Brown Blue Hazel Green
## Black 68 20 15 5
## Brown 119 84 54 29
## Red 26 17 14 14
## Blond 7 94 10 16
##
## $`Zeilenweise relative Häufigkeiten`
## Eye
## Hair Brown Blue Hazel Green
## Black 0.62962963 0.18518519 0.13888889 0.04629630
## Brown 0.41608392 0.29370629 0.18881119 0.10139860
## Red 0.36619718 0.23943662 0.19718310 0.19718310
## Blond 0.05511811 0.74015748 0.07874016 0.12598425
##
## $`Spaltenweise relative Häufigkeiten`
## Eye
## Hair Brown Blue Hazel Green
## Black 0.30909091 0.09302326 0.16129032 0.07812500
## Brown 0.54090909 0.39069767 0.58064516 0.45312500
## Red 0.11818182 0.07906977 0.15053763 0.21875000
## Blond 0.03181818 0.43720930 0.10752688 0.25000000##
## Call:
## CA(X = tab, row.sup = suprow, col.sup = supcol, graph = FALSE)
##
## The chi square of independence between the two variables is equal to 138.2898 (p-value = 2.325287e-25 ).
##
## Eigenvalues
## Dim.1 Dim.2 Dim.3
## Variance 0.209 0.022 0.003
## % of var. 89.373 9.515 1.112
## Cumulative % of var. 89.373 98.888 100.000
##
## Rows
## Iner*1000 Dim.1 ctr cos2 Dim.2 ctr cos2 Dim.3
## Black | 55.425 | 0.505 22.246 0.838 | -0.215 37.877 0.152 | -0.056
## Brown | 12.284 | 0.148 5.086 0.864 | 0.033 2.319 0.042 | 0.049
## Red | 15.095 | 0.130 0.964 0.133 | 0.320 55.131 0.812 | -0.083
## Blond | 150.793 | -0.835 71.704 0.993 | -0.070 4.673 0.007 | -0.016
## ctr cos2
## Black 21.633 0.010 |
## Brown 44.284 0.094 |
## Red 31.913 0.055 |
## Blond 2.171 0.000 |
##
## Columns
## Iner*1000 Dim.1 ctr cos2 Dim.2 ctr cos2 Dim.3
## Brown | 93.086 | 0.492 43.116 0.967 | -0.088 13.042 0.031 | -0.022
## Blue | 111.337 | -0.547 52.128 0.977 | -0.083 11.244 0.022 | 0.005
## Hazel | 13.089 | 0.213 3.401 0.542 | 0.167 19.804 0.336 | 0.101
## Green | 16.085 | -0.162 1.355 0.176 | 0.339 55.910 0.773 | -0.088
## ctr cos2
## Brown 6.680 0.002 |
## Blue 0.310 0.000 |
## Hazel 61.086 0.121 |
## Green 31.925 0.052 |Grafiken
library("factoextra")
fviz_ca(res, map="symmetric", arrows = c(FALSE, TRUE), repel = TRUE, title="Symmetrische Normalisierung")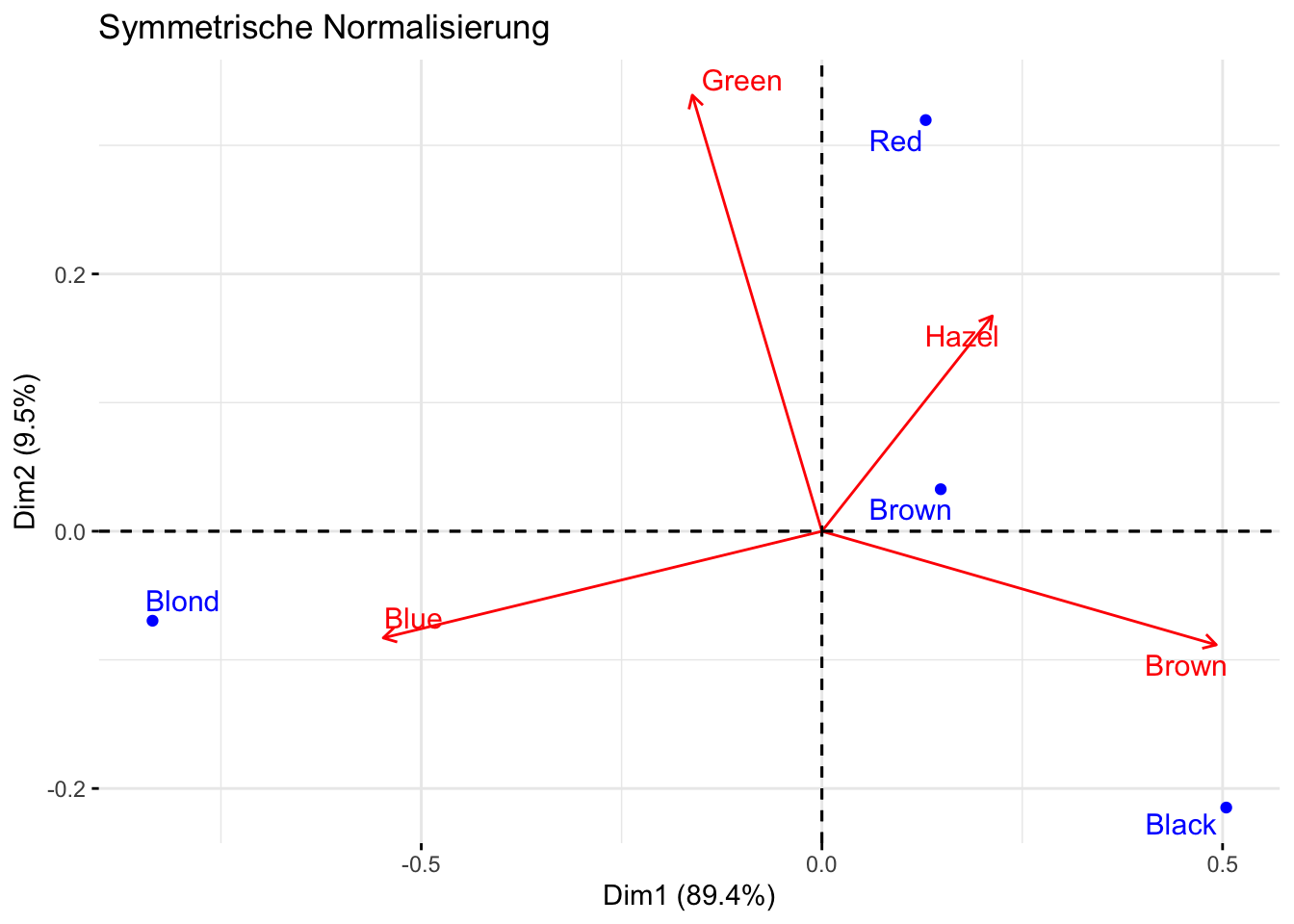
fviz_ca(res, map="rowprincipal", arrows = c(FALSE, TRUE), repel = TRUE, title="Zeilenprinzipal Normalisierung")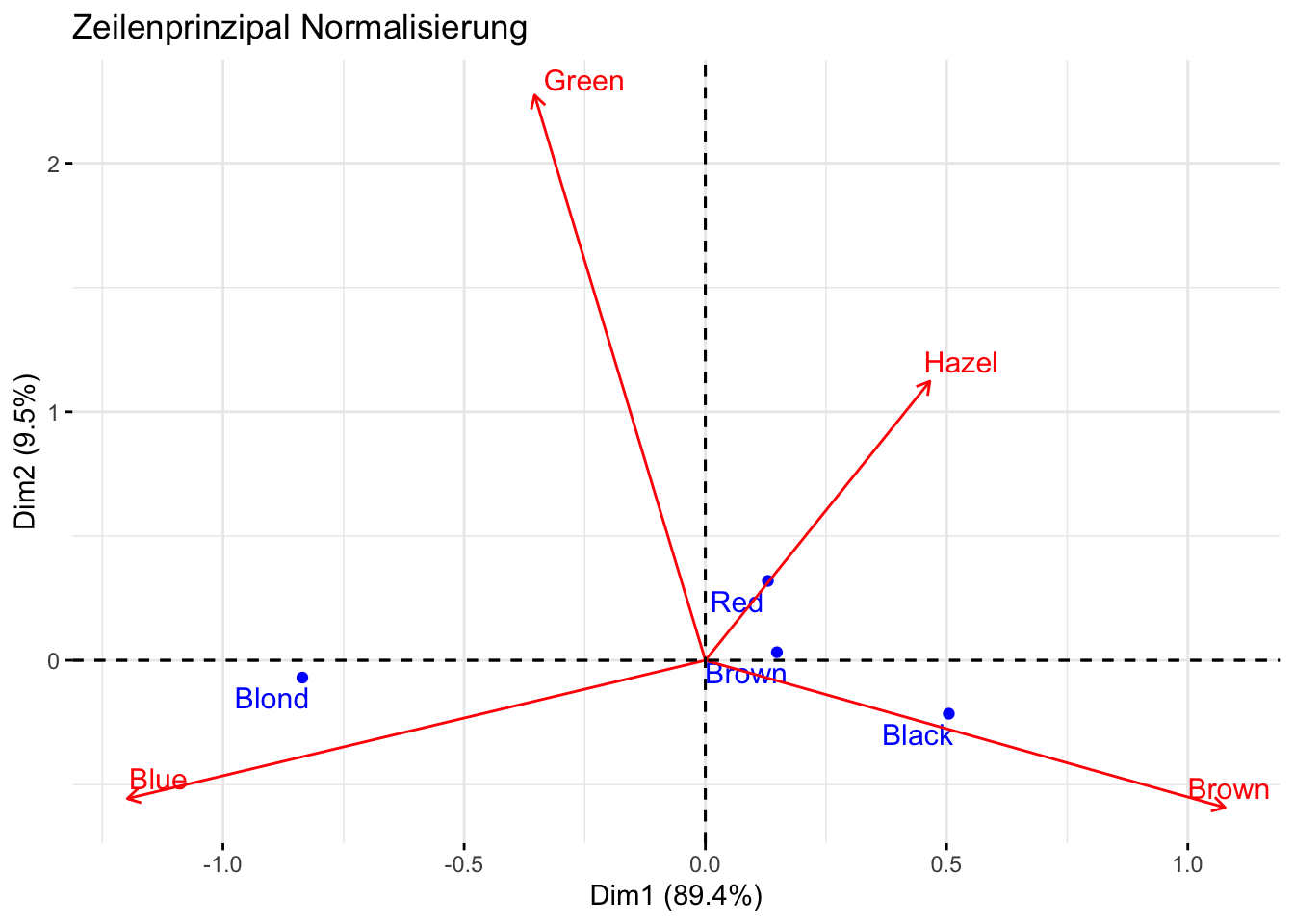
fviz_ca(res, map="colprincipal", arrows = c(FALSE, TRUE), repel = TRUE, title="Spaltenprinzipal Normalisierung")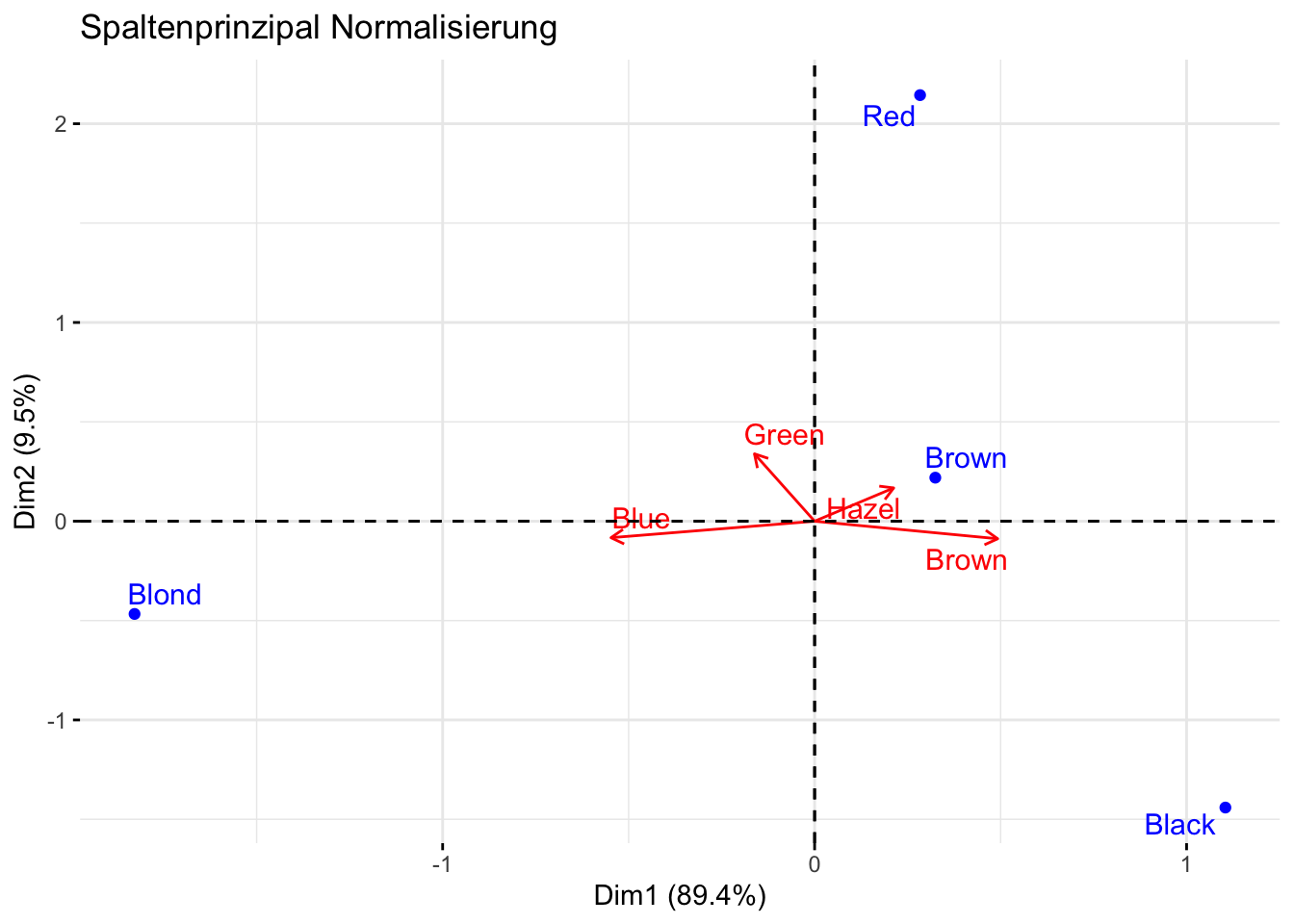
Hinweis: Weitere Plotvarianten vgl. http://www.sthda.com/english/wiki/print.php?id=228
Multiple Korrespondenzanalyse
Datensatz einlesen und Variablen spezifizieren
# Datensatz 'data': pro Versuchsperson eine Zeile
# Häufigkeitstabellen zuerst umwandeln, vgl. Umwandlung von Datenstrukturen
data(wg93)
data <- wg93
# Variablen spezifizieren
# Zusatzliche passive Variablen werden zu Vergleichzwecken in die Grafik eingefügt und beeinflussen
# die Berechnung derKorrespondenzanalyse nicht.
Variablen <- c(1:7) # Spaltennummern der aktiven und passiven Variablen
data2 <- data[,Variablen] # Datensatz data2 mit allen zu berücksichtigenden Variablen
quanti.sup <- NULL # Spaltennummern von data2 der quantitativen zusätzlichen passiven Variablen (keine=NULL)
quali.sup <- c(5:7) # Spaltennummern von data2 der qualitativen zusätzlichen passiven Variablen (keine=NULL)
ind.sup <- NULL # Zeilennummern der zusätzlichen passiven Zeilen bzw. Individuen (keine=NULL)Korrespondenzanalyse
# Alle direkt für die MCA verwendeten Variablen und die qualitativen zusätzlichen Variablen müssen Faktoren sein.
if (is.null(quanti.sup)) {NF <- 1:ncol(data2)} else {NF <- (1:ncol(data2))[-quanti.sup]} # Spaltennummern dieser Variablen
for (i in NF) data2[,i] <- as.factor(data2[,i]) # Diese Variablen als Faktoren definieren
res <- MCA(data2, quanti.sup=quanti.sup, quali.sup=quali.sup, ind.sup=ind.sup, method="Indicator", graph=FALSE); summary(res)##
## Call:
## MCA(X = data2, ind.sup = ind.sup, quanti.sup = quanti.sup, quali.sup = quali.sup,
## graph = FALSE, method = "Indicator")
##
##
## Eigenvalues
## Dim.1 Dim.2 Dim.3 Dim.4 Dim.5 Dim.6 Dim.7
## Variance 0.457 0.431 0.322 0.306 0.276 0.252 0.243
## % of var. 11.434 10.774 8.048 7.662 6.892 6.298 6.064
## Cumulative % of var. 11.434 22.209 30.257 37.919 44.810 51.109 57.173
## Dim.8 Dim.9 Dim.10 Dim.11 Dim.12 Dim.13 Dim.14
## Variance 0.235 0.225 0.221 0.210 0.197 0.178 0.169
## % of var. 5.874 5.637 5.516 5.246 4.929 4.447 4.228
## Cumulative % of var. 63.046 68.683 74.199 79.445 84.373 88.821 93.048
## Dim.15 Dim.16
## Variance 0.153 0.125
## % of var. 3.820 3.131
## Cumulative % of var. 96.869 100.000
##
## Individuals (the 10 first)
## Dim.1 ctr cos2 Dim.2 ctr cos2 Dim.3 ctr cos2
## 1 | -0.210 0.011 0.014 | -0.443 0.052 0.061 | 0.127 0.006 0.005 |
## 2 | -0.325 0.026 0.040 | -0.807 0.174 0.250 | 0.120 0.005 0.006 |
## 3 | 0.229 0.013 0.022 | -0.513 0.070 0.110 | -0.096 0.003 0.004 |
## 4 | 0.303 0.023 0.036 | -0.387 0.040 0.059 | 0.142 0.007 0.008 |
## 5 | -0.276 0.019 0.023 | -1.092 0.317 0.360 | 0.889 0.282 0.239 |
## 6 | -0.451 0.051 0.055 | 0.077 0.002 0.002 | -0.737 0.194 0.147 |
## 7 | -0.284 0.020 0.032 | -0.570 0.087 0.130 | -0.509 0.092 0.104 |
## 8 | -0.683 0.117 0.146 | -0.213 0.012 0.014 | -0.567 0.115 0.101 |
## 9 | 0.411 0.042 0.030 | -0.159 0.007 0.004 | 0.735 0.193 0.096 |
## 10 | -0.019 0.000 0.000 | -0.690 0.127 0.173 | 0.372 0.049 0.050 |
##
## Categories (the 10 first)
## Dim.1 ctr cos2 v.test Dim.2 ctr cos2 v.test
## A_1 | 1.242 11.522 0.244 14.574 | 0.478 1.808 0.036 5.603 |
## A_2 | 0.369 2.758 0.080 8.345 | -0.187 0.748 0.020 -4.218 |
## A_3 | -0.302 1.169 0.028 -4.929 | -0.787 8.424 0.190 -12.844 |
## A_4 | -0.788 6.945 0.160 -11.787 | 0.484 2.773 0.060 7.230 |
## A_5 | -1.349 5.485 0.106 -9.612 | 1.622 8.406 0.153 11.551 |
## B_1 | 1.978 17.427 0.347 17.378 | 0.899 3.825 0.072 7.903 |
## B_2 | 0.434 2.055 0.047 6.394 | -0.438 2.221 0.048 -6.452 |
## B_3 | 0.234 0.705 0.017 3.830 | -0.633 5.467 0.123 -10.355 |
## B_4 | -0.483 4.113 0.111 -9.831 | -0.184 0.633 0.016 -3.743 |
## B_5 | -0.916 7.364 0.161 -11.818 | 1.384 17.851 0.367 17.860 |
## Dim.3 ctr cos2 v.test
## A_1 -0.038 0.016 0.000 -0.452 |
## A_2 0.044 0.056 0.001 0.997 |
## A_3 0.315 1.808 0.030 5.143 |
## A_4 -0.821 10.699 0.173 -12.274 |
## A_5 1.504 9.685 0.132 10.716 |
## B_1 -0.300 0.571 0.008 -2.639 |
## B_2 0.231 0.826 0.013 3.401 |
## B_3 0.481 4.236 0.071 7.878 |
## B_4 -0.728 13.280 0.252 -14.820 |
## B_5 0.622 4.828 0.074 8.028 |
##
## Categorical variables (eta2)
## Dim.1 Dim.2 Dim.3
## A | 0.510 0.382 0.287 |
## B | 0.579 0.517 0.306 |
## C | 0.627 0.488 0.336 |
## D | 0.113 0.337 0.359 |
##
## Supplementary categories (the 10 first)
## Dim.1 cos2 v.test Dim.2 cos2 v.test Dim.3 cos2 v.test
## sex_1 | -0.143 0.020 -4.129 | -0.029 0.001 -0.847 | 0.020 0.000 0.572 |
## sex_2 | 0.137 0.020 4.129 | 0.028 0.001 0.847 | -0.019 0.000 -0.572 |
## age_1 | -0.166 0.003 -1.668 | 0.014 0.000 0.138 | -0.110 0.001 -1.110 |
## age_2 | -0.087 0.002 -1.451 | 0.081 0.002 1.353 | -0.072 0.002 -1.201 |
## age_3 | -0.025 0.000 -0.346 | 0.004 0.000 0.055 | 0.059 0.001 0.822 |
## age_4 | -0.031 0.000 -0.413 | -0.057 0.001 -0.761 | 0.074 0.001 0.974 |
## age_5 | 0.016 0.000 0.191 | -0.047 0.000 -0.566 | 0.010 0.000 0.124 |
## age_6 | 0.281 0.015 3.659 | -0.033 0.000 -0.434 | 0.027 0.000 0.351 |
## edu_1 | 0.180 0.001 1.134 | -0.060 0.000 -0.377 | 0.021 0.000 0.129 |
## edu_2 | 0.161 0.020 4.162 | -0.093 0.007 -2.413 | -0.001 0.000 -0.027 |
##
## Supplementary categorical variables (eta2)
## Dim.1 Dim.2 Dim.3
## sex | 0.020 0.001 0.000 |
## age | 0.018 0.003 0.004 |
## edu | 0.029 0.025 0.004 |Hinweis: Auswahlmöglichkeiten für Output
# resGrafiken
# Darstellung der Stufen der Spaltenelemente für die ersten beiden Dimensionen
fviz_mca_var(res, repel = TRUE, ggtheme = theme_minimal())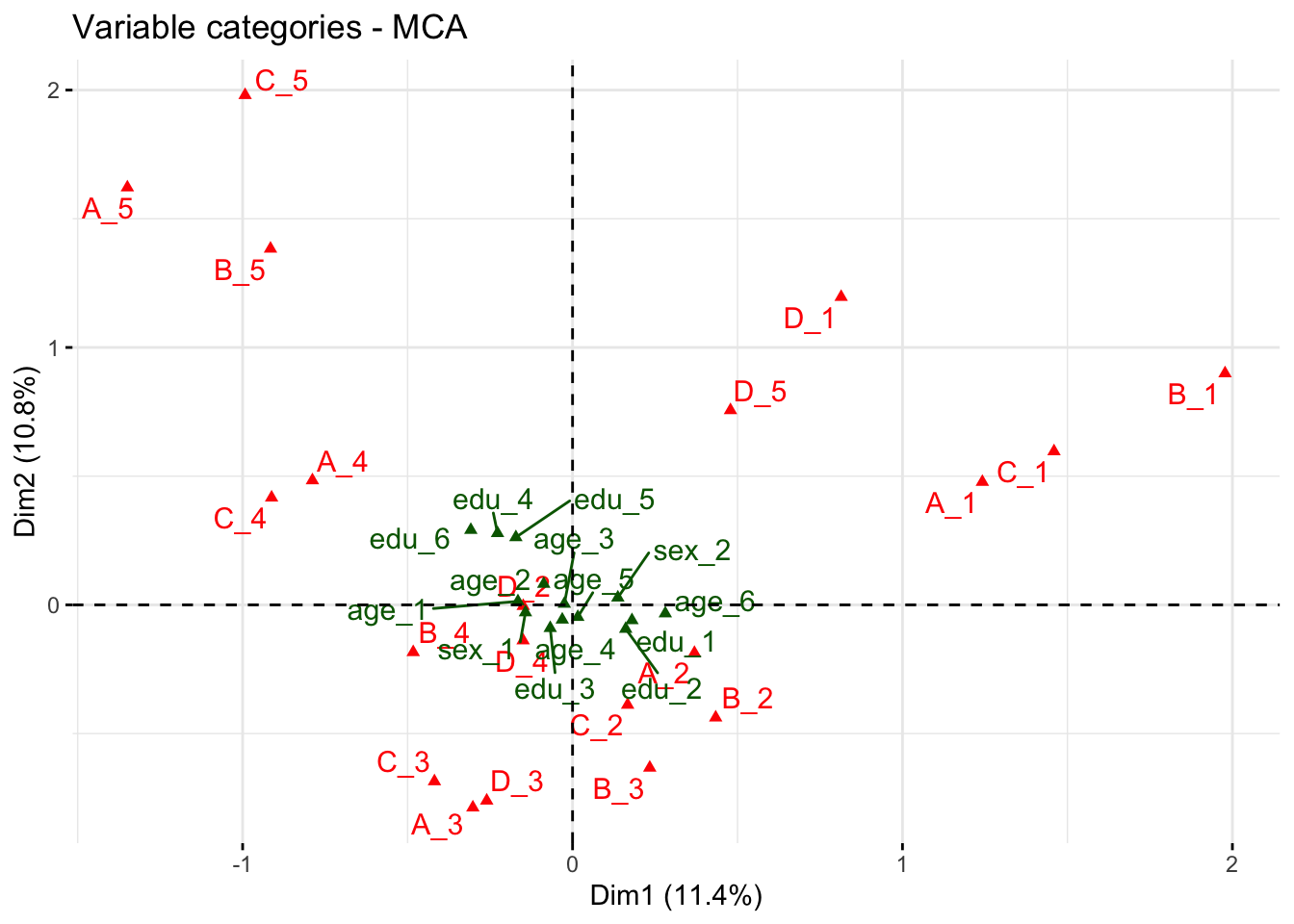
# Darstellung der Zeilenelemente für die ersten beiden Dimensionen
fviz_mca_ind(res, col.ind = "blue", geom="point")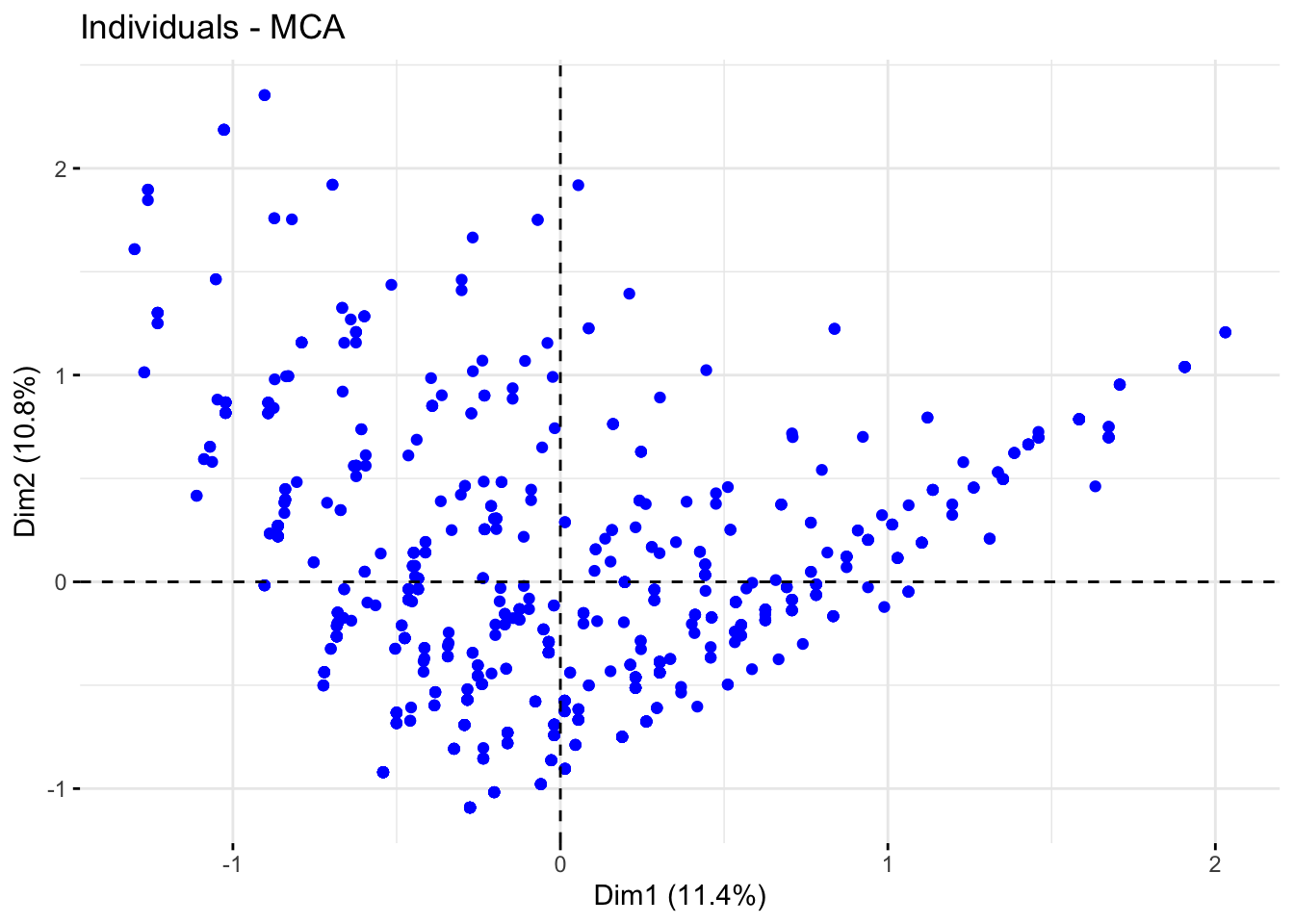
# Darstellung der erklärten Varianzanteile η2. Wichtigkeit der Faktoren für die Dimensionen
fviz_mca_var(res, choice = "mca.cor", repel = TRUE, ggtheme = theme_minimal())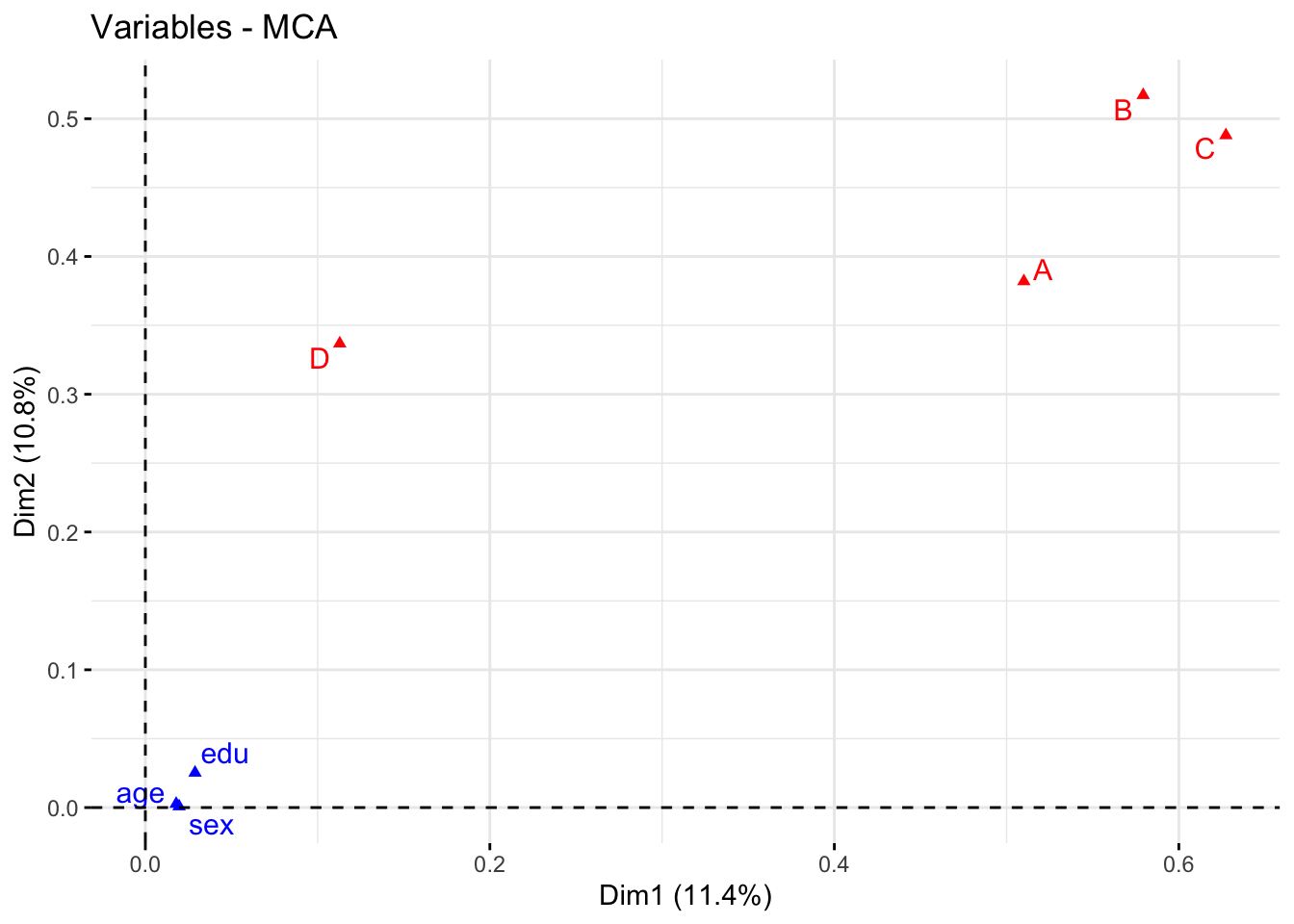
Hinweis: Weitere Plotvarianten http://www.sthda.com/english/articles/31-principal-component-methods-in-r-practical-guide/114-mca-multiple-correspondence-analysis-in-r-essentials/